- Beiträge: 1751
Sidebar
Identifizierung von diagnostischen Biomarkern für IPAH
12 Jan 2023 15:19 #1688
von danny
OMNIA TEMPUS HABENT
Diagnose IPAH im Februar 2013, in Behandlung bei OA Dr. Ulrich Krüger, jetzt Dr. Fischer Herzzentrum Duisburg, Medikamente: Sildenafil, Bosentan jetzt Macitentan, Subkutane Treprostinilpumpe, seit Januar 2024 getunnelter ZVK mit externer Pumpe (Groshongkatheter), 24/7 Sauerstoff, Marcumar, Diuretika
Identifizierung von diagnostischen Biomarkern für IPAH wurde erstellt von danny
Identifizierung von diagnostischen Biomarkern für idiopathische pulmonale Hypertonie mit metabolischem Syndrom durch Bioinformatik und maschinelles Lernen
www.nature.com/articles/s41598-023-27435-4
AbstraktIdiopathische pulmonale Hypertonie (IPAH) ist eine Erkrankung, die verschiedene Gewebe und Organe sowie das Stoffwechsel- und Entzündungssystem betrifft. Die am weitesten verbreitete Stoffwechselerkrankung ist das metabolische Syndrom (MS), das Insulinresistenz, Dyslipidämie und Fettleibigkeit umfasst. Basierend auf einer Fülle von Studien kann es einen Zusammenhang zwischen IPAH und MS geben, obwohl die zugrunde liegende Pathogenese unklar bleibt. Durch verschiedene bioinformatische Analysen und maschinelle Lernalgorithmen identifizierten wir 11 immun- und stoffwechselbezogene potenzielle diagnostische Gene ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1) für die Diagnose von IPAH und MS, und wir liefern hier ein Nomogramm für die Diagnose von IPAH bei MS-Patienten. Außerdem haben wir die abweichenden Immunzellen von IPAH entdeckt und diskutieren sie hier.EinführungDie pulmonale arterielle Hypertonie (PAH) ist eine seltene Erkrankung, die durch den Verschluss von Arteriolen in der Lunge gekennzeichnet ist, was zu einem deutlichen Anstieg des pulmonalen Gefäßwiderstands führt 1 . Es gibt viele Risikofaktoren für das Auftreten von PAH, darunter Stoffwechselstörungen, Hyperlipidämie, Fettleibigkeit, Insulinresistenz, fehlregulierte Proliferation von Gefäßzellen, abnormaler Zellstoffwechsel, Entzündungen und Genmutationen 2 . Idiopathische PAH (IPAH) ist eine wichtige Art von PAH, deren klinische Symptome nicht spezifisch sind; Die Patienten zeigen hauptsächlich Symptome im Zusammenhang mit einer fortschreitenden Funktionsstörung des rechten Herzens, die oft durch Müdigkeit verursacht wird, und zeigen Müdigkeit, Dyspnoe, Engegefühl in der Brust, Schmerzen in der Brust und Synkopen. IPAH wird durch mehrere pathogene Faktoren produziert, aber der spezifische pathogene Mechanismus wurde nicht vollständig aufgeklärt. Das metabolische Syndrom (MS) wird durch eine Reihe von kardiovaskulären und metabolischen Risikofaktoren ausgelöst, die miteinander in Verbindung stehen. Zu den Risikofaktoren gehören Stoffwechselstörungen, Bluthochdruck, Insulinresistenz, Glukoseintoleranz, zentrale Fettleibigkeit, Dyslipidämie und entzündliche Effekte 3 . Die Pathogenese der beiden Krankheiten weist Gemeinsamkeiten auf, und Patienten mit MS haben ein höheres Risiko für eine Anfälligkeit für IPAH.IPAH gilt als systemische Erkrankung und betrifft viele Organe und Gewebe sowie die Entzündungs- und Stoffwechselwege 4 . Die Rolle des knochenmorphogenen Proteinrezeptors Typ 2 (BMPR2) und vieler seiner nachgeschalteten Ziele, wie z. B. Peroxisom-Proliferator-aktivierter Rezeptor (PPAR)-γ und Apolipoprotein E, bei der Induktion der IPAH-Produktion über den Stoffwechselweg wurden ausführlich beschrieben, und PPAR -γ und Apolipoprotein E stehen ebenfalls in Zusammenhang mit einer Vielzahl von pathologischen Stoffwechselzuständen 5 . Stoffwechselstörungen sind seit langem bei IPAH weit verbreitet, und eine zunehmende Zahl von Studien deutet auf eine starke Verbindung zwischen IPAH und MS hin; Die beiden Krankheiten wurden jedoch nie zusammen untersucht. Daher wird diese Studie den Zusammenhang zwischen den beiden Krankheiten untersuchen und gemeinsame Biomarker für die Diagnose von Krankheiten aufdecken.Die bioinformatische Analyse hilft uns, die Ätiologie der Krankheit zu erforschen, während die Gen-Microarray-Technologie neue Ideen zur Erforschung der Pathogenese von IPAH und MS liefert. In dieser Studie haben wir maschinelle Lernalgorithmen für bioinformatische Analysen kombiniert, um diagnostische Kandidatengene und Signalwege zu identifizieren, die von IPAH und MS aus Gene Expression Omnibus-Datensätzen geteilt werden. Dies ist auch die erste Studie, die auf die gemeinsamen Biomarker und verwandte Stoffwechselwege von IPAH und MS abzielt, eine diagnostische Genexpressionsvalidierung wurde in einem anderen GEO-Datensatz durchgeführt. Unsere Studie liefert neue Erkenntnisse für die Erforschung der genetischen Ätiologie und Kombinationsbehandlungsstrategien für IPAH- und METS-Komorbidität. Darüber hinaus untersuchten wir auch die Infiltration von Immunzellen bei IPAH. Materialen und Methoden.DatenverarbeitungAus der Gene Expression Omnibus - Datenbank ( https : //www.ncbi.nlm .nih.gov/geo/ ) 9 . Die GSE15197-Serie umfasste Proben von 13 Kontrollgruppen und 18 IPAH-Patientengruppen und die GSE117261-Serie umfasste Proben von 20 Kontrollgruppen und 32 IPAH-Patientengruppen, während die GSE98895-Serie Proben von 20 Kontrollgruppen und 20 MS-Patientengruppen umfasste. Die IPAH-Datenbankproben wurden aus menschlichem Lungengewebe und die MS-Datenbankproben aus peripherem Blut von Patienten gewonnen. Ein einzelnes Validierungsset, GSE48149 10 , das 17 Lungengewebeproben von 8 IPAH-Patientengruppen und 9 normalen Kontrollen enthielt, wurde ebenfalls verwendet. Um Studienfehler zu vermeiden, haben wir Geschlecht und Alter zwischen Patienten und gesunden Kontrollen in den Datensätzen ausgeschlossen. Einzelheiten zu diesen Datensätzen sind in der Ergänzungstabelle S1 enthalten . Das Flussdiagramm dieser Studie ist in Abb. 1 dargestellt , gezeichnet vom WPS-Büro 11 (Kingsoft, China, Version: 11.1.0.11754).Abbildung 1
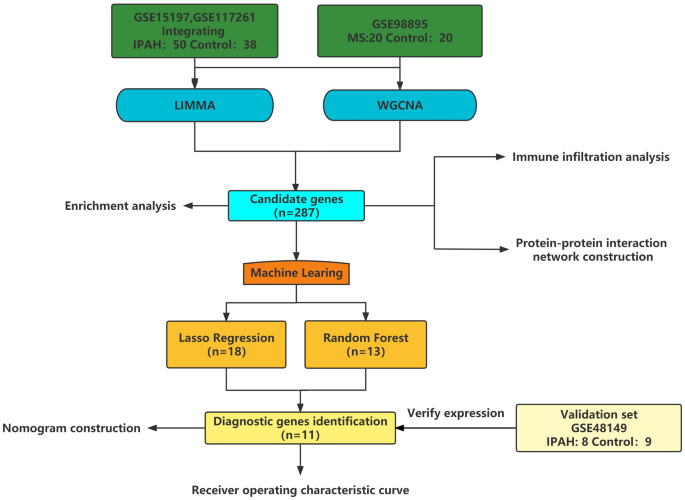 Flussdiagramm dieser Studie. Dawn von WPS-Büro (Version: 11.1.0.11754).
Screening auf differentiell exprimierte Gene (DEGs)Zunächst wurden beide IPAH-Rohdatensätze im Hintergrund kalibriert, normalisiert und log2-transformiert mit dem „affy“-Paket im R-Softwareprogramm
12
. Wenn mehrere Sonden identische Gene identifizierten, wurde der Mittelwert berechnet, um ihre Expression zu bestimmen. Für die Zusammenführung mehrerer Datenbanken haben wir zuerst die Datensätze mit R zusammengeführt, dann die Methode von Johnson, W. E verwendet, um Stapeleffekte zu entfernen, und schließlich die Matrix nach der Entfernung von Stapeleffekten erhalten
13
.Limma ist ein differentielles Expressions-Screening-Verfahren, das auf einem verallgemeinerten linearen Modell
14
basiert . Wir verwendeten das „limma“-Paket im R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
) für die Differentialanalyse, um die differentiellen Gene zwischen zu erhalten die verschiedenen Vergleichsgruppen und die Kontrollgruppen. Schließlich eine |log2-fache Änderung| Als Kriterien für die Identifizierung von DEGs mit dem „limma“-Paket wurden Werte von > 0,5 (IPAH-Filtration) bzw. 0,5 (MS-Filtration) und p < 0,05 festgelegt.Weighted Gene Co-Expression Network Analysis (WGCNA) und Modul-GenauswahlZuerst haben wir die mittlere absolute Abweichung für jedes Gen unter Verwendung des Genexpressionsmusters gezählt und dann die obersten 50 % der Gene mit minimaler mittlerer absoluter Abweichung entfernt. Die Funktion Good Samples Genes innerhalb von „WGCNA“ (Version 3.40.6; R Foundation for Statistical Computing, Vienna, Austria,
www.r-project.org/
) wurde verwendet, um nicht reservierte Gene zu entfernen
15
. Dann werden maßstabsfreie Koexpressionsnetzwerke aufgebaut. Die Pearson-Korrelationsmatrix und die Mean-Linkage-Methode wurden für alle gepaarten Genotypen verwendet. Wobei β der spezifizierte Soft-Schwellenwert-Parameter ist und die Potenzfunktion verwendet wird, um die gewichtete Adjazenzmatrix aufzubauen. Nach der Leistungsauswahl wird die Adjazenzbeziehung in eine topologische Überlappungsmatrix (TOM) transformiert und dann die entsprechende Anisotropie (1-TOM) berechnet. Darüber hinaus wird gemäß dem Anisotropiemaß von TOM die hierarchische Clusterbildung auf der durchschnittlichen Kette vervollständigt. Die Größe des Diagramms, das dem kleinsten Genbaum (Gencluster) entspricht, ist 100. Wir haben dann die Empfindlichkeit auf 3 gesetzt, Module mit einem Abstand < 0,25 kombiniert und schließlich 10 Module der Koexpression erhalten. Graue Module sind Sammlungen von Genen, die keinem Modul zuordenbar sind.Funktionelle AnreicherungsanalyseNach dem Screening dieser DEGs und WCGNA-Signatur-Biomarker führten wir eine Gene Ontology (GO) und eine Analyse der Kyoto Encyclopedia of Genomics (KEGG) durch. Die Funktionsanreicherungsanalyse von Gensätzen wurde unter Verwendung einer API im KEGG REST-Stil (
www.kegg.jp/kegg/rest/keggapi.html
) Software
16
durchgeführt . Genannotationen des jüngsten KEGG-Signalwegs als Hintergrund wurden erhalten und auf den Hintergrundsatz abgebildet; Die Anreicherungsanalyse wurde dann mit dem R-Paket „Profiler Cluster“ (Version 3.14.3) durchgeführt. Für die Zwecke dieser Analyse betrug die kleinste Kohortengröße 5 Gene und die größte Kohortengröße 5 000 Gene; Als statistisch signifikant wurden p < 0,05 und eine Fehlererkennungsrate < 0,1 angesehen.Wir haben auch das R-Paket „org.Hs.eg“ für GO-Annotationen verwendet. Wir führten eine GO-Annotation des Gens unter Verwendung von db (Version 3.1.0) als Hintergrund für die Anreicherungsanalyse durch und kartierten das Gen auf den Hintergrundsatz; Anschließend haben wir die Anreicherungsanalyse mit dem R-Paket „Cluster Profiler“ (Version 3.14.3) erneut ausgeführt, um das Ergebnis der Genset-Anreicherung zu erhalten. Die minimale Satzgröße betrug 5 Gene und die maximale Satzgröße 5.000 Gene; p < 0,05 und eine Falschentdeckungsrate < 0,1 wurden als statistisch signifikant angesehen.Aufbau von Protein-Protein-Wechselwirkungs(PPI)-NetzwerkenWir haben die STRING-Datenbank (Version 11.5;
www.string-db.org
) ausgewählt, um die Wechselwirkungen zwischen proteinkodierenden Genen
17
zu untersuchen , und wir haben ein PPI-Netzwerk eingerichtet. Die erforderliche minimale Interaktionspunktzahl betrug 0,4. Dann haben wir das Cytoscape-Softwareprogramm (Version 3.8.0;
www.cytoscape.org/
) verwendet, um die von STRING heruntergeladenen Bilder zu modifizieren und wichtige Interaktionsgene mit dem M-Code-Plugin
18
zu identifizieren .Algorithmen für maschinelles Lernen zum Screening nach diagnostischen KandidatengenenDer Random-Forest-Algorithmus und der Least-Absolute-Shrinkage-and-Selection-Operator (LASSO)-Algorithmus wurden verwendet, um nach diagnostischen Kandidatengenen am Schnittpunkt von DEGs und WGCNA-Modul-Genen zu suchen. Wir haben das „Random Forest“-R-Paket (Version 3.40.6) verwendet, um Random-Forest-Klassifikatoren zu erstellen, um Funktionen zu vergleichen und nach Wichtigkeit zu ordnen
19
. Dann haben wir mit dem R-Paket „glmnet“ (Version 3.40.6)
20
Genexpressionsdaten für die Regressionsanalyse mit der LASSO-Cox-Methode integriert. Darüber hinaus haben wir auch eine fünffache Kreuzvalidierung durchgeführt, um das optimale Modell zu erhalten. Durch diese beiden Algorithmen ausgewählte Gene wurden nacheinander als diagnostische Kandidatengene eingeschlossen.Nomogrammaufbau und Receiver Operating Characteristic (ROC)-KurvenauswertungUm die klinische Diagnose von IPAH zu erleichtern, konstruierten wir ein Nomogramm. Insbesondere haben wir auf der Grundlage der oben genannten diagnostischen Kandidatengene das „rms“ R-Paket (Version 3.40.6) verwendet, um das Nomogramm
21
zu erstellen . „Punkte“ gibt die Punktzahl der Kandidatengene an, und „Gesamtpunktzahl“ gibt die Summe aller Punktzahlen der oben genannten Gene an. Die Eichkurve des Nomogramms wurde ebenfalls erstellt. Anschließend wurde die ROC-Kurve erstellt, um den diagnostischen Wert der Kandidatengene zu bewerten, wonach die Fläche unter der ROC-Kurve (AUC) und die Werte des 95-%-Konfidenzintervalls (CI) berechnet wurden, um ihren Einfluss zu quantifizieren.statistische AnalyseDie Erstellung der ROC-Kurve und die Berechnung der AUC- und 95 %-KI-Werte wurden unter Verwendung von SPSS Version 26.0 (IBM Corporation, Armonk, NY, USA) abgeschlossen. Die Anteile verschiedener Immunzellen in den Kontroll- und IPAH-Gruppen wurden durch Anwenden des Student's t - Tests in GraphPad Prism Version 8.3.0 (Graph Pad Software, San Diego, CA, USA) verglichen. Wir betrachteten p < 0,05 als statistisch signifikant.Umfassende Korrelationsanalyse von infiltrierenden ImmunzellenIOBR ist ein Computertool für Studien zur Immuntumorbiologie
22
. CIBERSORT wurde basierend auf unseren Expressionsprofilen unter Verwendung des „IOBR“ R-Pakets (Version 3.40.6)
23
ausgewählt , und für jede Probe wurden 22 Werte für immuninfiltrierende Zellen berechnet. Der Anteil jedes Typs von Immunzellen in den verschiedenen Proben wurde mit Barplot visualisiert. VioPlot wurde verwendet, um den Vergleich unterschiedlicher Anteile von Typen von Immunzellen zwischen IPAH und Kontrollen zu visualisieren.
Mit dem „Corrplot“ R-Paket (Version 3.40.6) 24
wurde eine Heatmap erstellt, die die Korrelation der 22 Scores von infiltrierenden Immunzellen darstellt .Validierung diagnostischer KandidatengeneWie bereits erwähnt, haben wir den GSE48149-Datensatz, der 17 Lungengewebeproben mit 8 IPAH und 9 normalen Kontrollen enthält, als Validierungssatz für diese Studie ausgewählt. Und wir haben die Expression mehrerer diagnostischer Kandidatengene in diesem Datensatz analysiert.ErgebnisseDEGInsgesamt 159 DEGs wurden mit der Limma-Methode in der kombinierten IPAH-Datenbank identifiziert, von denen 88 erhöht und 71 herunterreguliert waren. Die Heatmap und die Vulkankarte der IPAH-DEGs sind in Abb.
2
A, C dargestellt. Für den MS-Datensatz wurden 1.467 DEGs (629 erhöht und 838 herunterreguliert) ausgewählt (Abb.
2
B, D). Der Schnittpunkt der beiden Gruppen von DEGs ist in Abb.
2
E dargestellt.Figur 2
Flussdiagramm dieser Studie. Dawn von WPS-Büro (Version: 11.1.0.11754).
Screening auf differentiell exprimierte Gene (DEGs)Zunächst wurden beide IPAH-Rohdatensätze im Hintergrund kalibriert, normalisiert und log2-transformiert mit dem „affy“-Paket im R-Softwareprogramm
12
. Wenn mehrere Sonden identische Gene identifizierten, wurde der Mittelwert berechnet, um ihre Expression zu bestimmen. Für die Zusammenführung mehrerer Datenbanken haben wir zuerst die Datensätze mit R zusammengeführt, dann die Methode von Johnson, W. E verwendet, um Stapeleffekte zu entfernen, und schließlich die Matrix nach der Entfernung von Stapeleffekten erhalten
13
.Limma ist ein differentielles Expressions-Screening-Verfahren, das auf einem verallgemeinerten linearen Modell
14
basiert . Wir verwendeten das „limma“-Paket im R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
) für die Differentialanalyse, um die differentiellen Gene zwischen zu erhalten die verschiedenen Vergleichsgruppen und die Kontrollgruppen. Schließlich eine |log2-fache Änderung| Als Kriterien für die Identifizierung von DEGs mit dem „limma“-Paket wurden Werte von > 0,5 (IPAH-Filtration) bzw. 0,5 (MS-Filtration) und p < 0,05 festgelegt.Weighted Gene Co-Expression Network Analysis (WGCNA) und Modul-GenauswahlZuerst haben wir die mittlere absolute Abweichung für jedes Gen unter Verwendung des Genexpressionsmusters gezählt und dann die obersten 50 % der Gene mit minimaler mittlerer absoluter Abweichung entfernt. Die Funktion Good Samples Genes innerhalb von „WGCNA“ (Version 3.40.6; R Foundation for Statistical Computing, Vienna, Austria,
www.r-project.org/
) wurde verwendet, um nicht reservierte Gene zu entfernen
15
. Dann werden maßstabsfreie Koexpressionsnetzwerke aufgebaut. Die Pearson-Korrelationsmatrix und die Mean-Linkage-Methode wurden für alle gepaarten Genotypen verwendet. Wobei β der spezifizierte Soft-Schwellenwert-Parameter ist und die Potenzfunktion verwendet wird, um die gewichtete Adjazenzmatrix aufzubauen. Nach der Leistungsauswahl wird die Adjazenzbeziehung in eine topologische Überlappungsmatrix (TOM) transformiert und dann die entsprechende Anisotropie (1-TOM) berechnet. Darüber hinaus wird gemäß dem Anisotropiemaß von TOM die hierarchische Clusterbildung auf der durchschnittlichen Kette vervollständigt. Die Größe des Diagramms, das dem kleinsten Genbaum (Gencluster) entspricht, ist 100. Wir haben dann die Empfindlichkeit auf 3 gesetzt, Module mit einem Abstand < 0,25 kombiniert und schließlich 10 Module der Koexpression erhalten. Graue Module sind Sammlungen von Genen, die keinem Modul zuordenbar sind.Funktionelle AnreicherungsanalyseNach dem Screening dieser DEGs und WCGNA-Signatur-Biomarker führten wir eine Gene Ontology (GO) und eine Analyse der Kyoto Encyclopedia of Genomics (KEGG) durch. Die Funktionsanreicherungsanalyse von Gensätzen wurde unter Verwendung einer API im KEGG REST-Stil (
www.kegg.jp/kegg/rest/keggapi.html
) Software
16
durchgeführt . Genannotationen des jüngsten KEGG-Signalwegs als Hintergrund wurden erhalten und auf den Hintergrundsatz abgebildet; Die Anreicherungsanalyse wurde dann mit dem R-Paket „Profiler Cluster“ (Version 3.14.3) durchgeführt. Für die Zwecke dieser Analyse betrug die kleinste Kohortengröße 5 Gene und die größte Kohortengröße 5 000 Gene; Als statistisch signifikant wurden p < 0,05 und eine Fehlererkennungsrate < 0,1 angesehen.Wir haben auch das R-Paket „org.Hs.eg“ für GO-Annotationen verwendet. Wir führten eine GO-Annotation des Gens unter Verwendung von db (Version 3.1.0) als Hintergrund für die Anreicherungsanalyse durch und kartierten das Gen auf den Hintergrundsatz; Anschließend haben wir die Anreicherungsanalyse mit dem R-Paket „Cluster Profiler“ (Version 3.14.3) erneut ausgeführt, um das Ergebnis der Genset-Anreicherung zu erhalten. Die minimale Satzgröße betrug 5 Gene und die maximale Satzgröße 5.000 Gene; p < 0,05 und eine Falschentdeckungsrate < 0,1 wurden als statistisch signifikant angesehen.Aufbau von Protein-Protein-Wechselwirkungs(PPI)-NetzwerkenWir haben die STRING-Datenbank (Version 11.5;
www.string-db.org
) ausgewählt, um die Wechselwirkungen zwischen proteinkodierenden Genen
17
zu untersuchen , und wir haben ein PPI-Netzwerk eingerichtet. Die erforderliche minimale Interaktionspunktzahl betrug 0,4. Dann haben wir das Cytoscape-Softwareprogramm (Version 3.8.0;
www.cytoscape.org/
) verwendet, um die von STRING heruntergeladenen Bilder zu modifizieren und wichtige Interaktionsgene mit dem M-Code-Plugin
18
zu identifizieren .Algorithmen für maschinelles Lernen zum Screening nach diagnostischen KandidatengenenDer Random-Forest-Algorithmus und der Least-Absolute-Shrinkage-and-Selection-Operator (LASSO)-Algorithmus wurden verwendet, um nach diagnostischen Kandidatengenen am Schnittpunkt von DEGs und WGCNA-Modul-Genen zu suchen. Wir haben das „Random Forest“-R-Paket (Version 3.40.6) verwendet, um Random-Forest-Klassifikatoren zu erstellen, um Funktionen zu vergleichen und nach Wichtigkeit zu ordnen
19
. Dann haben wir mit dem R-Paket „glmnet“ (Version 3.40.6)
20
Genexpressionsdaten für die Regressionsanalyse mit der LASSO-Cox-Methode integriert. Darüber hinaus haben wir auch eine fünffache Kreuzvalidierung durchgeführt, um das optimale Modell zu erhalten. Durch diese beiden Algorithmen ausgewählte Gene wurden nacheinander als diagnostische Kandidatengene eingeschlossen.Nomogrammaufbau und Receiver Operating Characteristic (ROC)-KurvenauswertungUm die klinische Diagnose von IPAH zu erleichtern, konstruierten wir ein Nomogramm. Insbesondere haben wir auf der Grundlage der oben genannten diagnostischen Kandidatengene das „rms“ R-Paket (Version 3.40.6) verwendet, um das Nomogramm
21
zu erstellen . „Punkte“ gibt die Punktzahl der Kandidatengene an, und „Gesamtpunktzahl“ gibt die Summe aller Punktzahlen der oben genannten Gene an. Die Eichkurve des Nomogramms wurde ebenfalls erstellt. Anschließend wurde die ROC-Kurve erstellt, um den diagnostischen Wert der Kandidatengene zu bewerten, wonach die Fläche unter der ROC-Kurve (AUC) und die Werte des 95-%-Konfidenzintervalls (CI) berechnet wurden, um ihren Einfluss zu quantifizieren.statistische AnalyseDie Erstellung der ROC-Kurve und die Berechnung der AUC- und 95 %-KI-Werte wurden unter Verwendung von SPSS Version 26.0 (IBM Corporation, Armonk, NY, USA) abgeschlossen. Die Anteile verschiedener Immunzellen in den Kontroll- und IPAH-Gruppen wurden durch Anwenden des Student's t - Tests in GraphPad Prism Version 8.3.0 (Graph Pad Software, San Diego, CA, USA) verglichen. Wir betrachteten p < 0,05 als statistisch signifikant.Umfassende Korrelationsanalyse von infiltrierenden ImmunzellenIOBR ist ein Computertool für Studien zur Immuntumorbiologie
22
. CIBERSORT wurde basierend auf unseren Expressionsprofilen unter Verwendung des „IOBR“ R-Pakets (Version 3.40.6)
23
ausgewählt , und für jede Probe wurden 22 Werte für immuninfiltrierende Zellen berechnet. Der Anteil jedes Typs von Immunzellen in den verschiedenen Proben wurde mit Barplot visualisiert. VioPlot wurde verwendet, um den Vergleich unterschiedlicher Anteile von Typen von Immunzellen zwischen IPAH und Kontrollen zu visualisieren.
Mit dem „Corrplot“ R-Paket (Version 3.40.6) 24
wurde eine Heatmap erstellt, die die Korrelation der 22 Scores von infiltrierenden Immunzellen darstellt .Validierung diagnostischer KandidatengeneWie bereits erwähnt, haben wir den GSE48149-Datensatz, der 17 Lungengewebeproben mit 8 IPAH und 9 normalen Kontrollen enthält, als Validierungssatz für diese Studie ausgewählt. Und wir haben die Expression mehrerer diagnostischer Kandidatengene in diesem Datensatz analysiert.ErgebnisseDEGInsgesamt 159 DEGs wurden mit der Limma-Methode in der kombinierten IPAH-Datenbank identifiziert, von denen 88 erhöht und 71 herunterreguliert waren. Die Heatmap und die Vulkankarte der IPAH-DEGs sind in Abb.
2
A, C dargestellt. Für den MS-Datensatz wurden 1.467 DEGs (629 erhöht und 838 herunterreguliert) ausgewählt (Abb.
2
B, D). Der Schnittpunkt der beiden Gruppen von DEGs ist in Abb.
2
E dargestellt.Figur 2
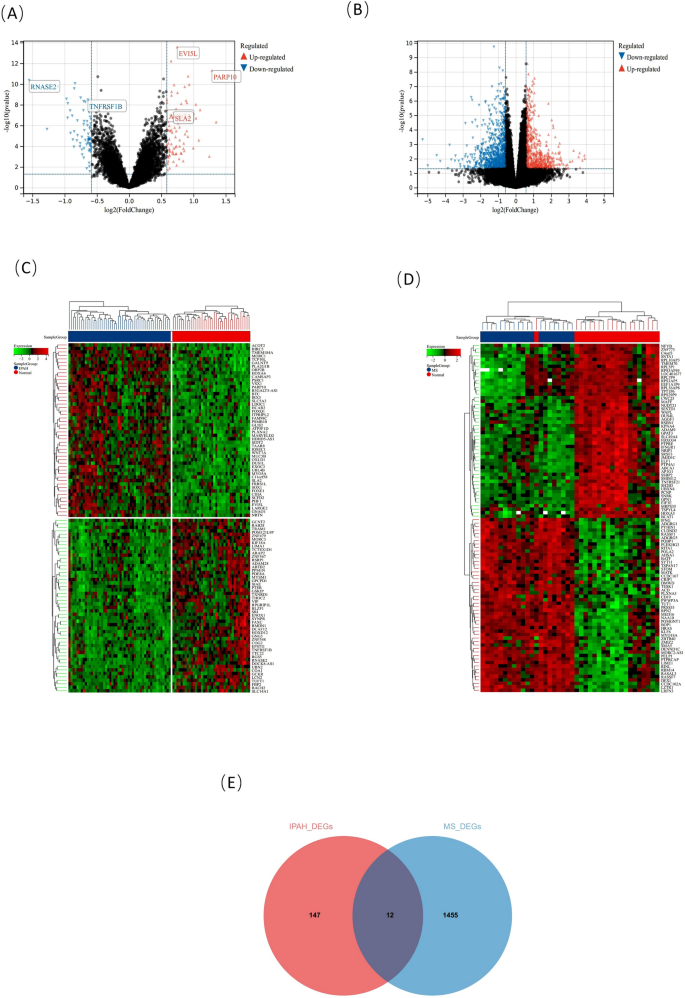 Vulkankarten und Heatmaps der IPAH- und MS-Datensätze. Gezeichnet durch das R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
). ( A ) Vulkankarte von DEGs im IPAH-Datensatz, |log2FC|> 0,5. ( B ) Vulkankarte von DEGs im MS-Datensatz, |log2FC|> 0,5. Rot steht für hochreguliert und Blau für herunterreguliert. ( C ) Heatmap von DEGs im IPAH-Datensatz. ( D ) Heatmap von DEGs im MS-Datensatz. Rot und Grün zeigen hochregulierte bzw. herunterregulierte DEGs an. ( E ) Venn-Plot überlappender IPAH- und MS-Datensätze DEGs.
WGCNA und SchlüsselmodulidentifikationWGCNA wurde verwendet, um die relevantesten Module in den IPAH- und MS-Gruppen zu identifizieren. In Bezug auf IPAH wurde β = 6 als weiche Schwelle basierend auf der Skalenunabhängigkeit und durchschnittlichen Konnektivität ausgewählt (Abb.
3
A), während β = 14 als weiche Schwelle für MS ausgewählt wurde (Abb.
3
Vulkankarten und Heatmaps der IPAH- und MS-Datensätze. Gezeichnet durch das R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
). ( A ) Vulkankarte von DEGs im IPAH-Datensatz, |log2FC|> 0,5. ( B ) Vulkankarte von DEGs im MS-Datensatz, |log2FC|> 0,5. Rot steht für hochreguliert und Blau für herunterreguliert. ( C ) Heatmap von DEGs im IPAH-Datensatz. ( D ) Heatmap von DEGs im MS-Datensatz. Rot und Grün zeigen hochregulierte bzw. herunterregulierte DEGs an. ( E ) Venn-Plot überlappender IPAH- und MS-Datensätze DEGs.
WGCNA und SchlüsselmodulidentifikationWGCNA wurde verwendet, um die relevantesten Module in den IPAH- und MS-Gruppen zu identifizieren. In Bezug auf IPAH wurde β = 6 als weiche Schwelle basierend auf der Skalenunabhängigkeit und durchschnittlichen Konnektivität ausgewählt (Abb.
3
A), während β = 14 als weiche Schwelle für MS ausgewählt wurde (Abb.
3
 . Abbildung
3
C,D zeigt das Clusterdendrogramm von IPAH/MS und Kontrollen. Basierend auf dieser Fähigkeit wurden in Bezug auf IPAH 15 Genkoexpressionsmodule generiert (Abb.
3
E, G). Eine Heatmap der Modulkorrelation mit Phänotypen ist in Abb.
4 dargestellt
A, wo die Türkis-, Cyan- und Lachs-Module (insgesamt 1168 Gene) die stärksten positiven Assoziationen mit IPAH hatten (R = 0,60, 0,42 und 0,45) und die Pink- und Lila-Module (insgesamt 612 Gene) die stärksten negativen Korrelationen mit IPAH aufwiesen IPAH (R = – 0,57 und – 0,39). In Bezug auf MS wurden basierend auf dieser Fähigkeit auch 10 Genkoexpressionsmodule generiert (Abb.
3
F, H), und eine Heatmap der Modulkorrelation mit dem Phänotyp ist in Abb.
4
B gezeigt, wo die magentafarbenen und gelben Module dargestellt sind (insgesamt 947 Gene) hatte die stärksten positiven Assoziationen mit MS (R = 0,71 und 0,74) und die braunen und rosa Module (insgesamt 2315 Gene) hatten die stärksten negativen Korrelationen mit MS (R = – 0,60 und – 0,53). Nach dem WGCNA-Screening erhielten wir 280 IPAH-Schnittpunkte mit MS-Modul-Genen (Abb.
4
C).Figur 3
. Abbildung
3
C,D zeigt das Clusterdendrogramm von IPAH/MS und Kontrollen. Basierend auf dieser Fähigkeit wurden in Bezug auf IPAH 15 Genkoexpressionsmodule generiert (Abb.
3
E, G). Eine Heatmap der Modulkorrelation mit Phänotypen ist in Abb.
4 dargestellt
A, wo die Türkis-, Cyan- und Lachs-Module (insgesamt 1168 Gene) die stärksten positiven Assoziationen mit IPAH hatten (R = 0,60, 0,42 und 0,45) und die Pink- und Lila-Module (insgesamt 612 Gene) die stärksten negativen Korrelationen mit IPAH aufwiesen IPAH (R = – 0,57 und – 0,39). In Bezug auf MS wurden basierend auf dieser Fähigkeit auch 10 Genkoexpressionsmodule generiert (Abb.
3
F, H), und eine Heatmap der Modulkorrelation mit dem Phänotyp ist in Abb.
4
B gezeigt, wo die magentafarbenen und gelben Module dargestellt sind (insgesamt 947 Gene) hatte die stärksten positiven Assoziationen mit MS (R = 0,71 und 0,74) und die braunen und rosa Module (insgesamt 2315 Gene) hatten die stärksten negativen Korrelationen mit MS (R = – 0,60 und – 0,53). Nach dem WGCNA-Screening erhielten wir 280 IPAH-Schnittpunkte mit MS-Modul-Genen (Abb.
4
C).Figur 3
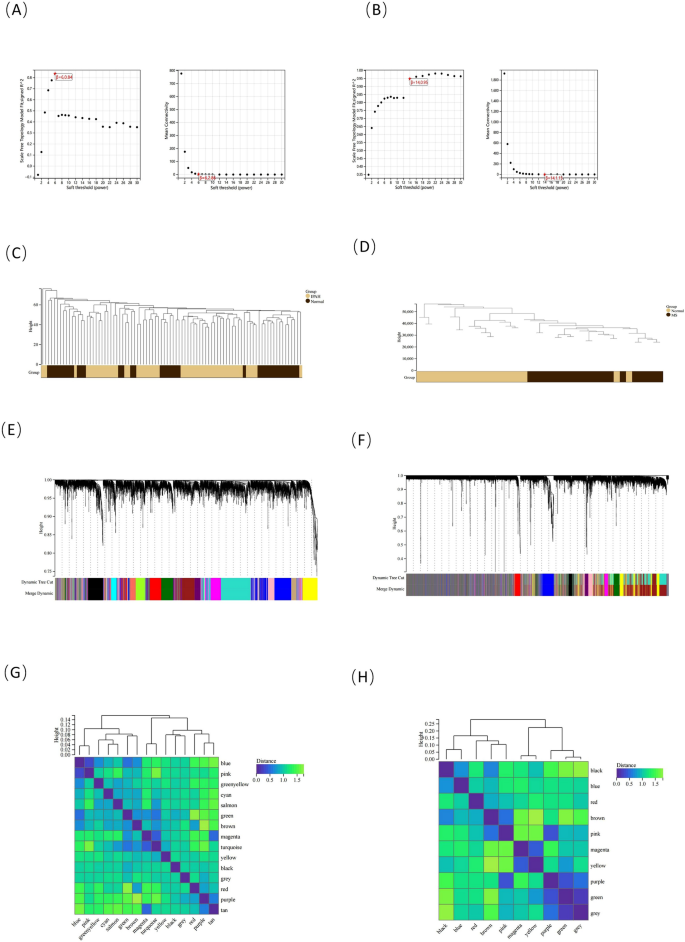 WGCNA bei IPAH und MS. Gezeichnet durch das R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
). ( A ) Analyse der Skalenunabhängigkeit und durchschnittlichen Konnektivität bei IPAH, weiche Schwelle β = 6 wählen. ( B ) Analyse der Skalenunabhängigkeit und durchschnittliche Konnektivität bei MS, weiche Schwelle β = 14 wählen. ( C, D ) Proben-Clustering basierend auf Expressionsniveau von Patienten im IPAH/MS-Datensatz. Ausreißerproben wurden gefiltert. ( E, F ) Unter dem Clustering-Baum werden Gen-Co-Expressionsmodule durch unterschiedliche Farben dargestellt. ( G,H ) Das IPAH/MS-Modul verfügt über eine Vektor-Clustering-Heatmap.
Figur 4
WGCNA bei IPAH und MS. Gezeichnet durch das R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
). ( A ) Analyse der Skalenunabhängigkeit und durchschnittlichen Konnektivität bei IPAH, weiche Schwelle β = 6 wählen. ( B ) Analyse der Skalenunabhängigkeit und durchschnittliche Konnektivität bei MS, weiche Schwelle β = 14 wählen. ( C, D ) Proben-Clustering basierend auf Expressionsniveau von Patienten im IPAH/MS-Datensatz. Ausreißerproben wurden gefiltert. ( E, F ) Unter dem Clustering-Baum werden Gen-Co-Expressionsmodule durch unterschiedliche Farben dargestellt. ( G,H ) Das IPAH/MS-Modul verfügt über eine Vektor-Clustering-Heatmap.
Figur 4
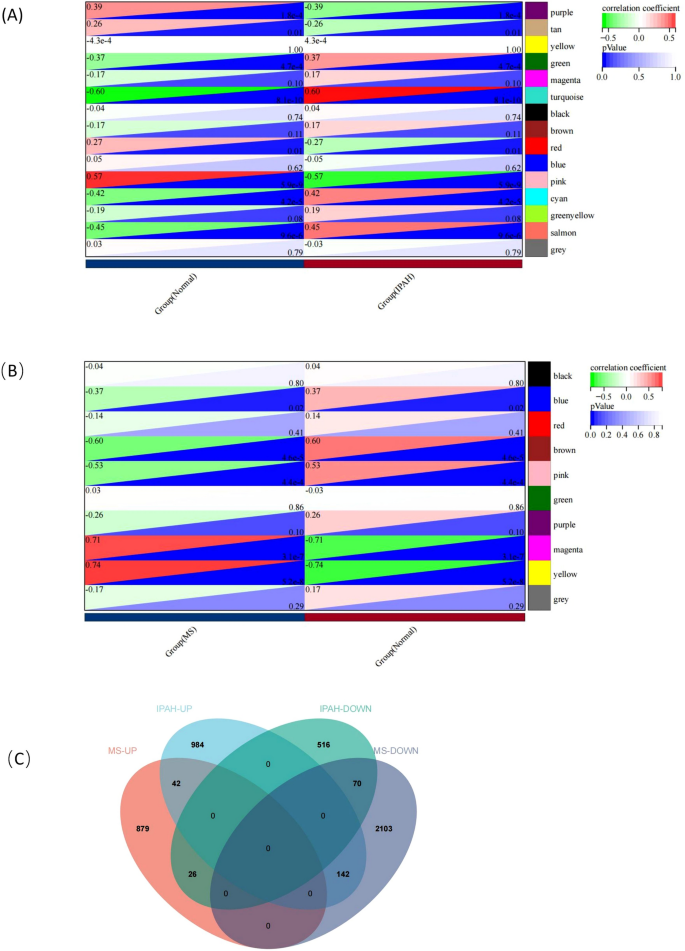 ( A ) Heatmap der Modulkorrelation mit IPAH. Türkis-, Cyan-, Lachs-Module hatten die stärkste positive Assoziation mit IPAH, rosa und violette Module hatten die stärkste negative Korrelation mit IPAH. ( B ) Heatmap der Modulkorrelation mit MS. Magentafarbene, gelbe Module hatten die stärkste positive Assoziation mit MS, braune und rosa Module hatten die stärkste negative Korrelation mit MS. ( C ) Venn-Plot überlappender Modulgene für IPAH und MS. Abbildung 4 wurde mit dem Softwareprogramm R (Version 3.40.6; R Foundation for Statistical Computing, Vienna, Austria,
www.r-project.org/
) gezeichnet.
Funktionelle AnreicherungsanalyseWir identifizierten insgesamt 12 Schnittgene in den IPAH- und MS-DEGs, darunter fünf Gene, die sich mit den von WGCNA ausgewählten Genen überschnitten. Um Auslassungen zu vermeiden, haben wir diese überlappenden Gene entfernt und die DEG-Gene und Modulgene als Kandidaten für die folgende Analyse kombiniert.Wir haben außerdem 287 Kandidatengene erhalten. Die KEGG-Analyse ergab, dass diese Gene hauptsächlich im „T-Zellrezeptor-Signalweg“ und im „Zentralen Kohlenstoffstoffwechsel bei Krebs“-Weg angereichert waren (Abb.
5
A). Die GO-Analyse ergab, dass die Gene hauptsächlich unter bestimmten biologischen Prozessbegriffen angereichert wurden, darunter „zellulärer Proteinstoffwechselprozess“ und „Immunsystemprozess“ (Abb.
5
( A ) Heatmap der Modulkorrelation mit IPAH. Türkis-, Cyan-, Lachs-Module hatten die stärkste positive Assoziation mit IPAH, rosa und violette Module hatten die stärkste negative Korrelation mit IPAH. ( B ) Heatmap der Modulkorrelation mit MS. Magentafarbene, gelbe Module hatten die stärkste positive Assoziation mit MS, braune und rosa Module hatten die stärkste negative Korrelation mit MS. ( C ) Venn-Plot überlappender Modulgene für IPAH und MS. Abbildung 4 wurde mit dem Softwareprogramm R (Version 3.40.6; R Foundation for Statistical Computing, Vienna, Austria,
www.r-project.org/
) gezeichnet.
Funktionelle AnreicherungsanalyseWir identifizierten insgesamt 12 Schnittgene in den IPAH- und MS-DEGs, darunter fünf Gene, die sich mit den von WGCNA ausgewählten Genen überschnitten. Um Auslassungen zu vermeiden, haben wir diese überlappenden Gene entfernt und die DEG-Gene und Modulgene als Kandidaten für die folgende Analyse kombiniert.Wir haben außerdem 287 Kandidatengene erhalten. Die KEGG-Analyse ergab, dass diese Gene hauptsächlich im „T-Zellrezeptor-Signalweg“ und im „Zentralen Kohlenstoffstoffwechsel bei Krebs“-Weg angereichert waren (Abb.
5
A). Die GO-Analyse ergab, dass die Gene hauptsächlich unter bestimmten biologischen Prozessbegriffen angereichert wurden, darunter „zellulärer Proteinstoffwechselprozess“ und „Immunsystemprozess“ (Abb.
5
 . In Bezug auf die Ontologie der zellulären Komponenten waren diese Gene hauptsächlich in den Kategorien „Zytosol“ und „Kernteil“ lokalisiert (Abb.
5
C). Molekulare Funktionsanalysen zeigten, dass „katalytische Aktivität“ und „katalytische Aktivität, die auf ein Protein wirkt“ die wichtigsten Kategorien unter den Genen waren (Abb.
5
D). Die Anreicherungsanalyse zeigte, dass die Kandidatengene hauptsächlich mit dem Metabolismus und der Immunantwort in Zusammenhang stehen und eng mit der Pathogenese von IPAH und MS verbunden sind, was starke Beweise für die nachfolgende Analyse liefert.Abbildung 5
. In Bezug auf die Ontologie der zellulären Komponenten waren diese Gene hauptsächlich in den Kategorien „Zytosol“ und „Kernteil“ lokalisiert (Abb.
5
C). Molekulare Funktionsanalysen zeigten, dass „katalytische Aktivität“ und „katalytische Aktivität, die auf ein Protein wirkt“ die wichtigsten Kategorien unter den Genen waren (Abb.
5
D). Die Anreicherungsanalyse zeigte, dass die Kandidatengene hauptsächlich mit dem Metabolismus und der Immunantwort in Zusammenhang stehen und eng mit der Pathogenese von IPAH und MS verbunden sind, was starke Beweise für die nachfolgende Analyse liefert.Abbildung 5
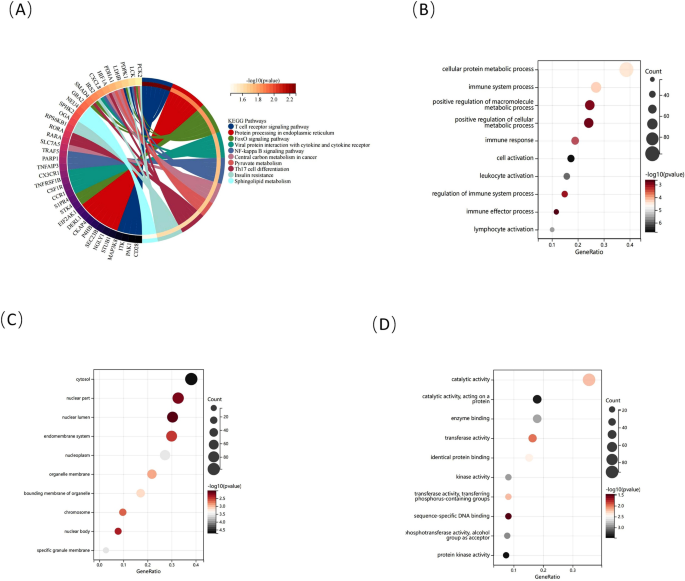 Anreicherungsanalyse von Kandidatengenen. ( A ) Die KEGG-Weganalyse ist in einem Kreisdiagramm dargestellt, wobei unterschiedliche Farben unterschiedliche Wege darstellen. ( B–D ) Go-Analyse von Kandidatengenen, einschließlich biologischer Prozesse, zellulärer Komponenten und molekularer Funktionen. Eingefärbt nach P-Werten stellt die X-Achse den Anteil angereicherter Gene dar und die Y-Achse unterschiedliche Ergebnisse.
PPI-NetzwerkaufbauNachdem wir bestätigt hatten, dass die ausgewählten Gene eng mit Immunität und Stoffwechsel zusammenhängen, bauten wir ein PPI-Netzwerk auf, um Knotengene zu identifizieren. Abbildung
6
A zeigt das PPI-Netzwerk, in dem die aktivsten Module mithilfe des M-Code-Plugins für Cytoscape (Abb.
6
Anreicherungsanalyse von Kandidatengenen. ( A ) Die KEGG-Weganalyse ist in einem Kreisdiagramm dargestellt, wobei unterschiedliche Farben unterschiedliche Wege darstellen. ( B–D ) Go-Analyse von Kandidatengenen, einschließlich biologischer Prozesse, zellulärer Komponenten und molekularer Funktionen. Eingefärbt nach P-Werten stellt die X-Achse den Anteil angereicherter Gene dar und die Y-Achse unterschiedliche Ergebnisse.
PPI-NetzwerkaufbauNachdem wir bestätigt hatten, dass die ausgewählten Gene eng mit Immunität und Stoffwechsel zusammenhängen, bauten wir ein PPI-Netzwerk auf, um Knotengene zu identifizieren. Abbildung
6
A zeigt das PPI-Netzwerk, in dem die aktivsten Module mithilfe des M-Code-Plugins für Cytoscape (Abb.
6
 visualisiert wurden. 13 Gene wurden als Hub-Gene identifiziert, und es wurde festgestellt, dass die funktionelle Anreicherung hauptsächlich im „Immunsystemprozess“ und im „Zelloberflächenrezeptor-Signalweg“ angereichert ist. Dies zeigt, dass die Hub-Gene eher eine zentrale Rolle im PPI-Netzwerk durch das Immunsystem spielen. Spezifische Informationen sind in der Ergänzungstabelle
S2
dargestellt .Abbildung 6
visualisiert wurden. 13 Gene wurden als Hub-Gene identifiziert, und es wurde festgestellt, dass die funktionelle Anreicherung hauptsächlich im „Immunsystemprozess“ und im „Zelloberflächenrezeptor-Signalweg“ angereichert ist. Dies zeigt, dass die Hub-Gene eher eine zentrale Rolle im PPI-Netzwerk durch das Immunsystem spielen. Spezifische Informationen sind in der Ergänzungstabelle
S2
dargestellt .Abbildung 6
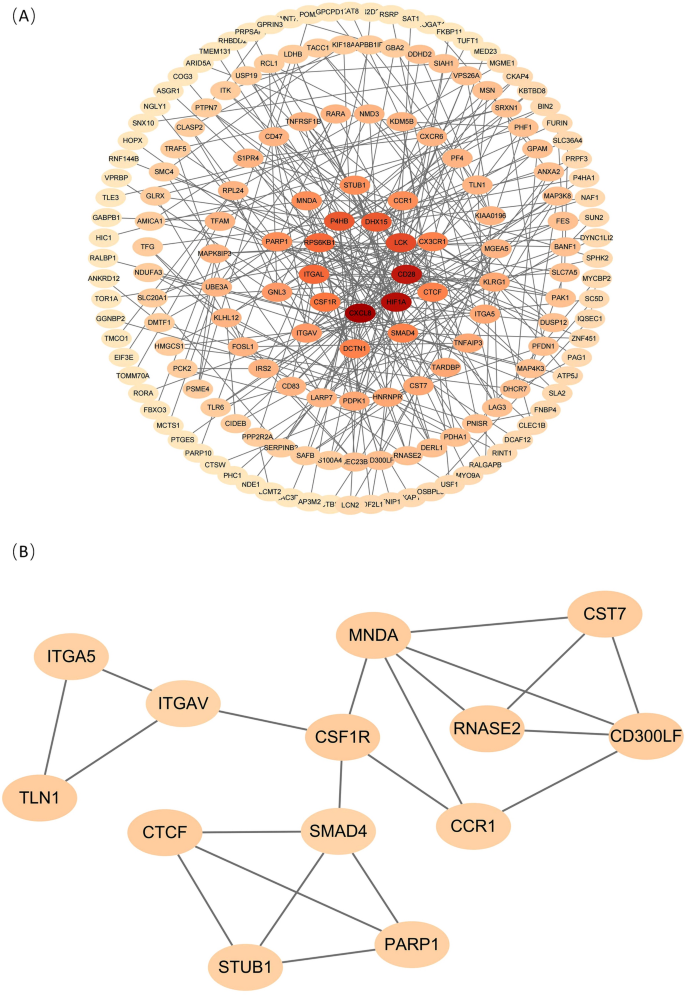 PPI-Netzwerk. Gezeichnet von der STRING-Datenbank (Version 11.5;
www.string-db.org
) und der Cytoscape-Software (Version 3.8.0;
www.cytoscape.org/
). ( A ) PPI-Netzwerk von Kandidatengenen. Unterschiedliche Genfarben zeigen den Kerngrad des Gens im PPI-Netzwerk an, und je dunkler die Farbe, desto höher der Kerngrad. ( B ) Filtern Sie die bekanntesten Module nach dem MCODE-Plugin.
Identifizierung diagnostischer Kandidatengene durch maschinelles LernenIn dieser Studie haben wir die LASSO-Regression und den Random-Forest-Machine-Learning-Algorithmus angewendet, um Kandidatengene für die Nomogrammkonstruktion und die Bewertung des diagnostischen Werts zu filtern. (Abb.
7
A,
PPI-Netzwerk. Gezeichnet von der STRING-Datenbank (Version 11.5;
www.string-db.org
) und der Cytoscape-Software (Version 3.8.0;
www.cytoscape.org/
). ( A ) PPI-Netzwerk von Kandidatengenen. Unterschiedliche Genfarben zeigen den Kerngrad des Gens im PPI-Netzwerk an, und je dunkler die Farbe, desto höher der Kerngrad. ( B ) Filtern Sie die bekanntesten Module nach dem MCODE-Plugin.
Identifizierung diagnostischer Kandidatengene durch maschinelles LernenIn dieser Studie haben wir die LASSO-Regression und den Random-Forest-Machine-Learning-Algorithmus angewendet, um Kandidatengene für die Nomogrammkonstruktion und die Bewertung des diagnostischen Werts zu filtern. (Abb.
7
A,  zeigt, dass der LASSO-Regressionsalgorithmus 18 potenzielle Biomarkerkandidaten identifizierte und der Algorithmus im Random Forest die Gene nach Wichtigkeit ordnete (Abb.
7
C, D). Mit Hilfe eines Venn-Diagramms (Abb.
7
E) zeigen wir die Schnittmenge der 16 höchstrangigen Gene aus der Stichprobe des Waldes mit den Top 18 potenziellen LASSO-Kandidatengenen und den Top 11 Genen ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1 ) können als den höchsten diagnostischen Wert angesehen werden.Abbildung 7
zeigt, dass der LASSO-Regressionsalgorithmus 18 potenzielle Biomarkerkandidaten identifizierte und der Algorithmus im Random Forest die Gene nach Wichtigkeit ordnete (Abb.
7
C, D). Mit Hilfe eines Venn-Diagramms (Abb.
7
E) zeigen wir die Schnittmenge der 16 höchstrangigen Gene aus der Stichprobe des Waldes mit den Top 18 potenziellen LASSO-Kandidatengenen und den Top 11 Genen ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1 ) können als den höchsten diagnostischen Wert angesehen werden.Abbildung 7
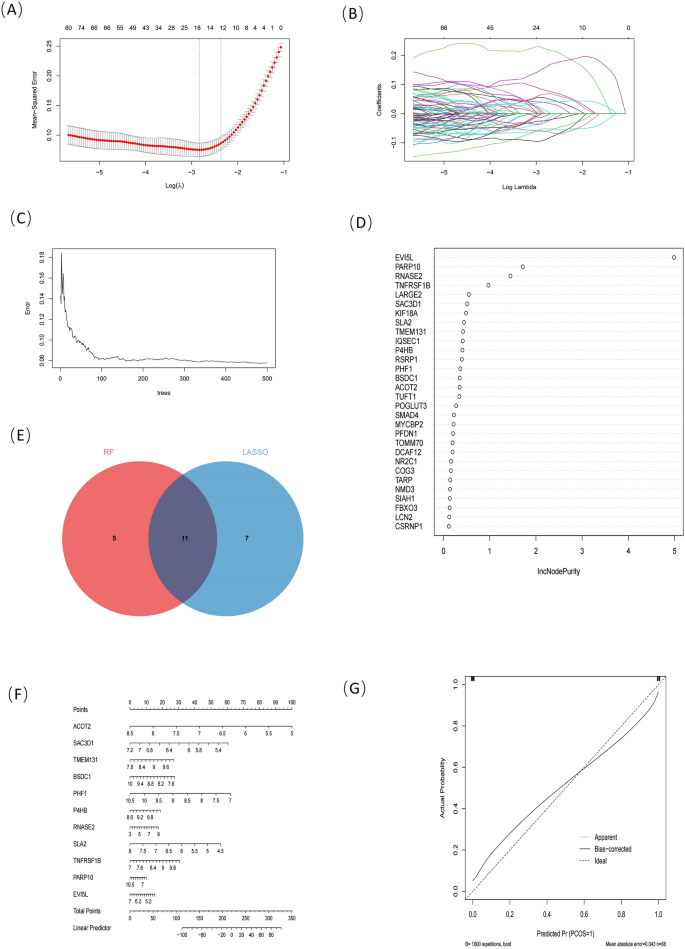 Maschinelles Lernen. Gezeichnet durch das R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
). ( A,B ) Gene werden durch den LASSO-Algorithmus gescreent. Um das optimale Modell zu erhalten, wird die Methode der zehnfachen Kreuzvalidierung angewendet. Die niedrigste Gennummer n = 18 am tiefsten Punkt der Kurve ist am besten für LASSO geeignet. ( C,D ) Screenen von Genen über Random-Forest-Algorithmus. Die Top 30 signifikanten Gene, die von Random Forest erkannt wurden. IncNodePurity ordnet die Gene nach ihrer relativen Bedeutung. ( E ) Venn-Diagramm der Schnittmenge zweier Algorithmen. ( F ) Nomogramm für IPAH- und MS-Diagnostik. ( G) Kalibrierungskurve des Nomogramms, nahe der Diagonale zeigt eine hohe Genauigkeit an.
Diagnostische WertschätzungBasierend auf den 11 diagnostischen Kandidatengenen wurde ein Nomogramm konstruiert (Fig.
7F
). Es wurden auch ROC-Kurven erstellt, um die diagnostische Spezifität und Sensitivität jedes Gens und Nomogramms zu beurteilen. Die Kalibrierungskurve des Nomogramms ist in Abb.
7
G dargestellt, und je näher die Bias-korrigierte Linie an der Diagonale liegt, desto größer ist der diagnostische Wert des Nomogramms. Wir haben die AUC- und 95 %-KI-Werte für jedes Projekt berechnet. Drei Gene mit den höchsten AUC-Werten wurden für die Anzeige in Abb.
8
A–C wie folgt ausgewählt: EVI5L (AUC = 0,95, 95 % CI 0,91–0,99), RNASE2 (AUC = 0,89, 95 % CI 0,82–0,96), und PARP10(AUC = 0,88, 95 %-KI 0,80–0,95). Die AUC-Werte für alle Gene sind in der Ergänzungstabelle
S3
gezeigt . Alle Kandidatengene hatten einen diagnostischen Wert für IPAH bei MS; andere Gene hatten AUC-Werte, die zwischen 0,76 und 0,88 schwankten.Abbildung 8
Maschinelles Lernen. Gezeichnet durch das R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing, Wien, Österreich,
www.r-project.org/
). ( A,B ) Gene werden durch den LASSO-Algorithmus gescreent. Um das optimale Modell zu erhalten, wird die Methode der zehnfachen Kreuzvalidierung angewendet. Die niedrigste Gennummer n = 18 am tiefsten Punkt der Kurve ist am besten für LASSO geeignet. ( C,D ) Screenen von Genen über Random-Forest-Algorithmus. Die Top 30 signifikanten Gene, die von Random Forest erkannt wurden. IncNodePurity ordnet die Gene nach ihrer relativen Bedeutung. ( E ) Venn-Diagramm der Schnittmenge zweier Algorithmen. ( F ) Nomogramm für IPAH- und MS-Diagnostik. ( G) Kalibrierungskurve des Nomogramms, nahe der Diagonale zeigt eine hohe Genauigkeit an.
Diagnostische WertschätzungBasierend auf den 11 diagnostischen Kandidatengenen wurde ein Nomogramm konstruiert (Fig.
7F
). Es wurden auch ROC-Kurven erstellt, um die diagnostische Spezifität und Sensitivität jedes Gens und Nomogramms zu beurteilen. Die Kalibrierungskurve des Nomogramms ist in Abb.
7
G dargestellt, und je näher die Bias-korrigierte Linie an der Diagonale liegt, desto größer ist der diagnostische Wert des Nomogramms. Wir haben die AUC- und 95 %-KI-Werte für jedes Projekt berechnet. Drei Gene mit den höchsten AUC-Werten wurden für die Anzeige in Abb.
8
A–C wie folgt ausgewählt: EVI5L (AUC = 0,95, 95 % CI 0,91–0,99), RNASE2 (AUC = 0,89, 95 % CI 0,82–0,96), und PARP10(AUC = 0,88, 95 %-KI 0,80–0,95). Die AUC-Werte für alle Gene sind in der Ergänzungstabelle
S3
gezeigt . Alle Kandidatengene hatten einen diagnostischen Wert für IPAH bei MS; andere Gene hatten AUC-Werte, die zwischen 0,76 und 0,88 schwankten.Abbildung 8
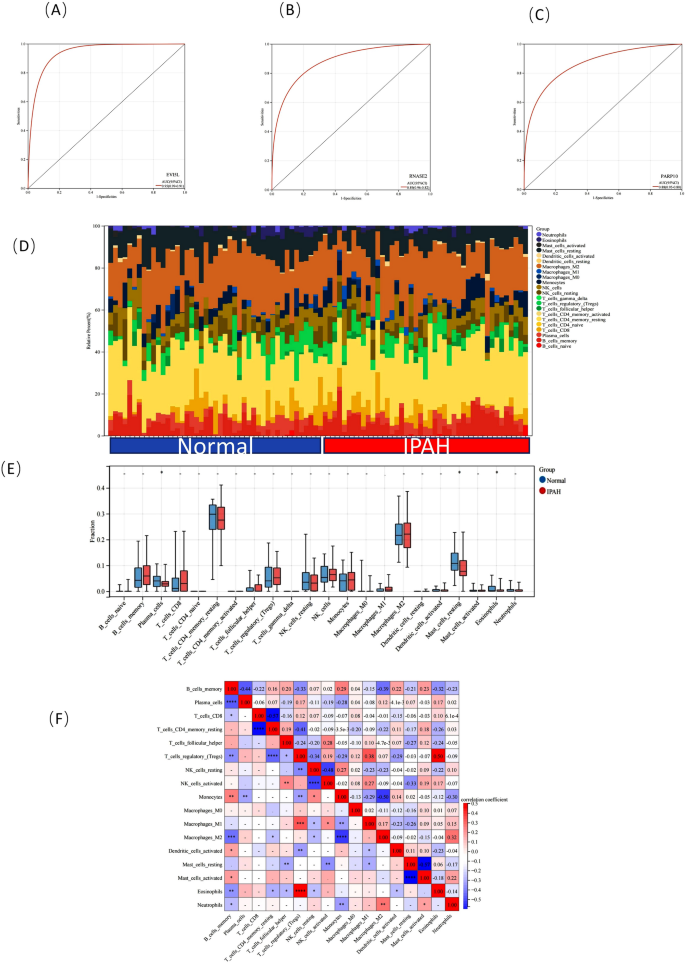 ( A–C ) ROC-Kurve eines Teils der diagnostischen Kandidatengene: (EVI5L (AUC = 0,95,95 % CI 0,91–0,99), RNASE2 (AUC = 0,89,95 % CI 0,82–0,96), PARP10 (AUC = 0,88, 95 % KI 0,80–0,95).( D ) Für jede Probe wurden zweiundzwanzig Werte für immuninfiltrierende Zellen berechnet.Verwendung von Barplot, um den Anteil jedes Typs von Immunzellen in verschiedenenProben sichtbar zu machen.( E ) Vergleich von Immunzellen mit unterschiedlichen Proportionen zwischen IPAH- und Kontrollgruppen. ( F ) IPAH-Immunzellverhältnis-bezogene Heatmap. Rot stellt eine positive Korrelation dar und Blau stellt eine negative Korrelation dar. Abbildung 8 wird mit dem R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing) gezeichnet , Wien, Österreich,
www.r-project.org/
).
ImmunzellinfiltrationsanalyseBasierend auf den Ergebnissen früherer Anreicherungsanalysen kann gefolgert werden, dass die gemeinsamen Gene von IPAH und MS in metabolischen und immunbezogenen Signalwegen angereichert sind und als diagnostisches Mittel für die potenzielle Biomarkerhäufigkeit von IPAH verwendet werden können. Daher können die relevanten Mechanismen durch die Immunzellinfiltrationsanalyse von IPAH besser erforscht werden. Für die IPAH- und Kontrollgruppen ist der Anteil von 22 Immunzellen in jeder Probe in Abb.
8
D dargestellt. Abbildung
8
E zeigt, dass IPAH-Patienten höhere Konzentrationen an Gedächtnis-B-Zellen, CD8-T-Zellen, follikulären Helfer-T-Zellen, Monozyten und M1- und M2-Makrophagen und niedrigere Konzentrationen an Plasmazellen, gedächtnisruhenden CD4-T-Zellen und regulatorischen T-Zellen aufweisen (Tregs), ruhende natürliche Killerzellen (NK), NK-Zellen, ruhende Mastzellen und Eosinophile. Die Korrelation von 22 Arten von Immunzellen ergab, dass Tregs positiv mit Eosinophilen (r = 0,50) und Makrophagen M1 (r = 0,38) assoziiert waren, während CD8-T-Zellen negativ mit CD4-T-Zellen im Gedächtnis ruhten (r = – 0,57). ) (Abb.
8
F). Bei IPAH-Patienten unterschieden sich verschiedene Immunzellinfiltrate, was ein potenzieller regulatorischer Punkt für die IPAH-Behandlung sein könnte.Validierung diagnostischer KandidatengeneUm die diagnostische Kandidatengenexpression bei IPAH-Patienten zu validieren, analysierten wir die Expression der differenziellen Gene im Validierungssatz, und die Korrelationsergebnisse sind in
9
gezeigt . Die Ergebnisse zeigten, dass die diagnostischen Kandidatengene im Lungengewebedatensatz von IPAH-Patienten unterschiedlich exprimiert wurden, wobei der Unterschied in RNASE2 am signifikantesten war.Abbildung 9
( A–C ) ROC-Kurve eines Teils der diagnostischen Kandidatengene: (EVI5L (AUC = 0,95,95 % CI 0,91–0,99), RNASE2 (AUC = 0,89,95 % CI 0,82–0,96), PARP10 (AUC = 0,88, 95 % KI 0,80–0,95).( D ) Für jede Probe wurden zweiundzwanzig Werte für immuninfiltrierende Zellen berechnet.Verwendung von Barplot, um den Anteil jedes Typs von Immunzellen in verschiedenenProben sichtbar zu machen.( E ) Vergleich von Immunzellen mit unterschiedlichen Proportionen zwischen IPAH- und Kontrollgruppen. ( F ) IPAH-Immunzellverhältnis-bezogene Heatmap. Rot stellt eine positive Korrelation dar und Blau stellt eine negative Korrelation dar. Abbildung 8 wird mit dem R-Softwareprogramm (Version 3.40.6; R Foundation for Statistical Computing) gezeichnet , Wien, Österreich,
www.r-project.org/
).
ImmunzellinfiltrationsanalyseBasierend auf den Ergebnissen früherer Anreicherungsanalysen kann gefolgert werden, dass die gemeinsamen Gene von IPAH und MS in metabolischen und immunbezogenen Signalwegen angereichert sind und als diagnostisches Mittel für die potenzielle Biomarkerhäufigkeit von IPAH verwendet werden können. Daher können die relevanten Mechanismen durch die Immunzellinfiltrationsanalyse von IPAH besser erforscht werden. Für die IPAH- und Kontrollgruppen ist der Anteil von 22 Immunzellen in jeder Probe in Abb.
8
D dargestellt. Abbildung
8
E zeigt, dass IPAH-Patienten höhere Konzentrationen an Gedächtnis-B-Zellen, CD8-T-Zellen, follikulären Helfer-T-Zellen, Monozyten und M1- und M2-Makrophagen und niedrigere Konzentrationen an Plasmazellen, gedächtnisruhenden CD4-T-Zellen und regulatorischen T-Zellen aufweisen (Tregs), ruhende natürliche Killerzellen (NK), NK-Zellen, ruhende Mastzellen und Eosinophile. Die Korrelation von 22 Arten von Immunzellen ergab, dass Tregs positiv mit Eosinophilen (r = 0,50) und Makrophagen M1 (r = 0,38) assoziiert waren, während CD8-T-Zellen negativ mit CD4-T-Zellen im Gedächtnis ruhten (r = – 0,57). ) (Abb.
8
F). Bei IPAH-Patienten unterschieden sich verschiedene Immunzellinfiltrate, was ein potenzieller regulatorischer Punkt für die IPAH-Behandlung sein könnte.Validierung diagnostischer KandidatengeneUm die diagnostische Kandidatengenexpression bei IPAH-Patienten zu validieren, analysierten wir die Expression der differenziellen Gene im Validierungssatz, und die Korrelationsergebnisse sind in
9
gezeigt . Die Ergebnisse zeigten, dass die diagnostischen Kandidatengene im Lungengewebedatensatz von IPAH-Patienten unterschiedlich exprimiert wurden, wobei der Unterschied in RNASE2 am signifikantesten war.Abbildung 9
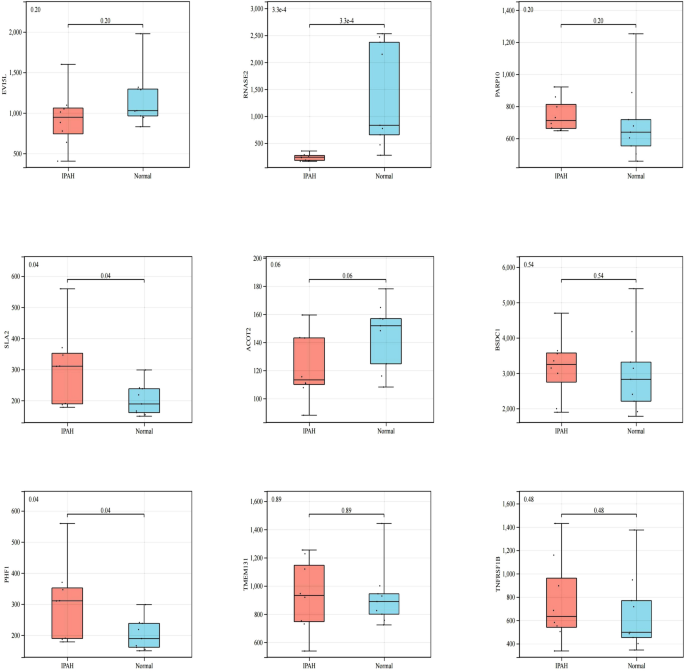 Der IPAH-Datensatz GSE48149 für Lungengewebe verifiziert die Expression diagnostischer Kandidatengene.
DiskussionDie Ätiologie der IPAH ist unbekannt, dennoch stellt die Krankheit eine große physische, mentale und wirtschaftliche Belastung für die Patienten dar. Bestehende Studien haben einen Teil neuer Biomarker identifiziert, die die Diagnose von IPAH erleichtern können. Über Immuninfiltrationsstudien von IPAH wurde ursprünglich bereits berichtet, aber diese Untersuchung ist die erste, die IPAH mit MS kombiniert. In der Zwischenzeit wurde die Identifizierung von diagnostischen Kandidatengenen bei der Diagnose von IPAH nicht berücksichtigt. Wir haben eine Reihe integrierter bioinformatischer Analysen und maschineller Lernmethoden verwendet, um gemeinsame Signalwege und gemeinsame diagnostische Kandidatengene für IPAH und MS zu identifizieren. Um Fehler zu vermeiden, haben wir die DEGs- und WGCNA-Modulgene kombiniert, um insgesamt 287 gemeinsame Kandidatengene zu identifizieren. Anreicherungsanalysen zeigten, dass diese Kandidatengene mit immun- und stoffwechselbezogenen Signalwegen assoziiert sind. Als nächstes wendeten wir einen maschinellen Lernansatz an, um weiter nach Schlüsselgenen zu suchen. Die Crossover-Ergebnisse von Random-Forest- und LASSO-Analysen wurden als gemeinsame diagnostische Kandidatengene für IPAH und MS betrachtet, und wir haben die diagnostische Wirkung jedes gemeinsamen diagnostischen Kandidatengens weiter validiert. Insbesondere,EVI5L , RNASE2 und PARP10 haben einen großen diagnostischen Wert und hohe AUC-Werte.IPAH ist eine seltene Erkrankung, die durch einen erhöhten Lungengefäßwiderstand gekennzeichnet ist. In dieser Studie haben wir zwei Datensätze mit IPAH-Lungengewebe als Analyseproben ausgewählt, die für die Genexpression bei IPAH-Patienten repräsentativer waren als die Gensequenzierung von peripherem Blut. Wir haben dann die obigen Ergebnisse anhand der Daten verifiziert und festgestellt, dass die identifizierten diagnostischen Kandidatengene in einem anderen IPAH-Lungengewebedatensatz gleichermaßen unterschiedlich exprimiert wurden. Daher könnten wir schlussfolgern, dass die entdeckten diagnostischen Kandidatengene versteckte IPAH durch periphere Blutuntersuchungen von MS-Patienten erkennen können, was eine wahnsinnig einfache und wirtschaftliche Operation ist und eine invasive Untersuchung durch Rechtsherzkatheter vermeidet.Letztendlich identifizierten wir 11 wichtige Kandidatengene ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1 ) und es wurde auch ein Nomogramm zur Diagnose von IPAH bei MS-Patienten erstellt, das einen hohen diagnostischen Wert aufweist.EVI5L gehört zu einer kleinen Unterfamilie von TRE-2/BUB2/CDC16-Domänenproteinen und ist ein Nebenprodukt von EVI5 . EVI5L hat etwa eine zu 70 % ähnliche Identität wie Evi5 . Aufgrund der wenigen vorliegenden Berichte über EVI5L haben wir jedoch hauptsächlich EVI5 analysiert. EVI5 hat verschiedene regulatorische Rollen bei der Zellzyklusprogression, der Zytokinese und dem Transport von Zellmembranen. In Tumoren ist die EVI5- Expression bei mehreren Krebsarten wie nicht-kleinzelligem Lungenkrebs, Kehlkopfkrebs und hepatozellulärem Karzinom fehlreguliert, und EVI5 gilt daher als potenzielle Onkogene und Zellzyklusregulatoren
25
,
26
,
27
.EVI5 ist auch ein Risikofaktor für Multiple Sklerose
28
. Multiple Sklerose ist eine ziemlich häufige demyelinisierende Autoimmunerkrankung; EVI5L könnte daher als immunbezogenes Gen eine wichtige Rolle bei der zellulären Immunität spielen. Der Mechanismus von EVI5L bei IPAH und MS erfordert jedoch weitere Untersuchungen.RNASE2 ist ein von Eosinophilen abgeleitetes Neurotoxin (EDN/RNase2) und eine endolysosomale Ribonuklease, die synergistisch wirkt, um Uridin aus Oligonukleotiden freizusetzen. RNASE2 aktiviert den humanen Toll-like-Rezeptor 8 (TLR8), während die TLR8-Aktivierung eine starke T-Helfer-1-Zellantwort induziert, die für die Abwehr intrazellulärer Pathogene entscheidend ist. Dies deutet darauf hin, dass RNASE2 eine wichtige Rolle im Immunsystem spielt
29
. Als immunbezogenes Molekül ist RNASE2 ein Biomarker für verschiedene Erkrankungen des Immunsystems, darunter chronische myeloische Leukämie, systemischer Lupus erythematodes, rheumatoide Arthritis und multiples Myelom
30
,
31
,
32
,
33
. In Bezug auf Krebs fördert RNASE2 die maligne Progression von Gliomen über den PI3K/Akt-Signalweg
34
. Es ist auch ein immunbezogener Biomarker, der zur Bewertung der Prognose von Magen- und Nierenkrebs verwendet wird
35
,
36
. Im Atmungssystem beeinflusst RNASE2 die Eosinophilen-spezifischen Proteinspiegel der Asthmafamilie und spielt eine Schlüsselrolle bei allergischen Reaktionen, die Asthma auslösen
37
. Frühere bioinformatische Studien haben gezeigt, dass RNASE2 in IPAH überexprimiert wird und ein Biomarker von IPAH ist
38
. Die bestehende Forschung definiert jedoch die Hauptrolle von RNASE2 noch nicht klarim IPAH. In dieser Studie fanden wir heraus, dass RNASE2 ein gemeinsamer immun- und stoffwechselbezogener Biomarker für MS und IPAH ist, was darauf hindeutet, dass RNASE2 für die Entwicklung von Stoffwechselstörungen bei beiden Krankheiten verantwortlich sein könnte, was beweist, dass es eine wichtige potenzielle Rolle bei der Diagnose spielt MS-Patienten mit IPAH.PARP10 , alternativ bekannt als ARTD10 , ist ein Mitglied der PARP-Proteinfamilie, das eine Mono-ADP-Ribosylierung von Zielproteinen durchführt
39
. PARP10 ist ein Stoffwechselregulator, der eine wichtige Rolle im Fettstoffwechsel spielt. Das Silencing von PARP10 induziert mitochondriale Oxidation und AMPK-Aktivität. PARP10 ist an der Regulierung der zellulären Autophagie in Zellmodellen beteiligt; in einem Zellkrebsmodell induziert der Verlust von PARP10 die Fettsäureoxidation
40
. PARP10 wird häufig in menschlichen Geweben exprimiert, insbesondere in Leber und Milz. Die Sekretion von Apolipoprotein B in der Leber ist abhängig von PARP10 , undDie Stummschaltung von PARP10 reduziert die Expression von Apolipoprotein B in menschlichen Hepatozyten
41
. Daher kann die Expression von PARP10 Lipoproteinspiegel sehr niedriger, mittlerer Dichte und niedriger Dichte beeinflussen, und PARP10 ist eng mit dem Lipidstoffwechsel verbunden. PARP10 ist auch an der Entzündungsreaktion und der Tumorentwicklung beteiligt, da es in den meisten menschlichen Tumoren überexprimiert wird, einschließlich Brust- und Eierstocktumoren, oralem Plattenepithelkarzinom, kolorektalem Karzinom und hepatozellulärem Karzinom, und PARP10 spielt auch eine Rolle bei der Förderung der Proliferation von verwandte Tumoren
42
,
43
,
44
,
45
. Darüber hinaus ist PARP10 für Anti-DNA-Schäden erforderlich, und der Knockout des PARP10 -Gens verursacht eine zelluläre Überempfindlichkeit gegenüber DNA-Schäden und einen DNA-Replikationsdefekt
46
. Wir stellten fest, dass die Crossover-Gene von IPAH und MS hauptsächlich in Stoffwechsel- und Immunwegen angereichert sind und stellten fest, dass PARP10 als Stoffwechselregulator eine wichtige Rolle bei der Entstehung und Entwicklung beider Krankheiten spielt. Unsere Studie hat gezeigt, dass die Überexpression von PARP10 bei Patienten mit IPAH mit MS ein lebenswichtiger stoffwechselbezogener Biomarker bei Patienten sein kann und einen hohen diagnostischen Wert hat.Stoffwechselstörungen sind eine wichtige Pathogenese von PAH, und die medikamentöse Behandlung des pathologischen Stoffwechselzustands eines Patienten zur Behandlung von erhöhtem Lungenarteriendruck ist ein Bereich, der von vielen Forschern aktiv untersucht wird. Tiermodelltests ergaben, dass das hypoglykämische Medikament Metformin die Endothelfunktion bei PAH verbesserte und das Überleben von PAH-Ratten signifikant verlängerte
47
. Die Ergebnisse einer klinischen Studie bestätigten auch, dass Biguanid, ein oral verabreichtes hypoglykämisches Medikament, die rechtsventrikuläre Fraktionsfläche von PAH-Patienten signifikant verbesserte, mit einer guten therapeutischen Wirkung
48
. Legtschenko et al. fanden heraus, dass der PPAR-γ-Agonist Pioglitazon die pulmonale Hypertonie durch Fettsäureoxidation umkehrte, die hauptsächlich mit dem Fettstoffwechsel und einer gestörten mitochondrialen Morphologie/Funktion bei rechtsventrikulärem Versagen und pulmonalvaskulärer Hypertonie verbunden war
49
. Der Natrium-Glucose-Cotransporter 2 (SGLT2)-Inhibitor Englizin verbesserte die Glukoseausscheidung im Urin und reduzierte kardiovaskuläre Ereignisse und Mortalität bei Patienten mit Typ-2-Diabetes. In ihrer Studie
50
stellten sie fest, dass SGLT2 die Sterblichkeit bei MCT-induzierten PAH-Ratten reduzierte und den maladaptiven Lungenumbau reduzierte.Entzündung ist eine kritische Komponente aller Subtypen von PAH, aktivierte Immunzellen sezernierten mehr Zytokine, wie z. B. Tumornekrosefaktor-α und Interleukine, und können in den Seren von Patienten in Konzentrationen gefunden werden, die positiv mit der Schwere der Erkrankung bei PAH korrelieren
51
. Es wurde gezeigt, dass viele zirkulierende Immunzellen (z. B. Makrophagen, Monozyten, Mastzellen, dendritische Zellen und T-Zellen) bei PAH in Milz und Lunge aktiviert werden, und eine große Anzahl wird in den Lungenkreislauf rekrutiert oder innerhalb des Lungenkreislaufs aktiviert. Sie regulieren die Funktion und den Differenzierungsstatus der Pulmonalarterienzellen auf parakrine Weise. Die Arten von Immunzellen, die an PAH beteiligt sind, können bei Aktivierung stark glykolytisch werden, was darauf hindeutet, dass diese Zellen möglicherweise auch auf veränderte Stoffwechseltherapien und andere Faktoren ansprechen
51
. Frühere Untersuchungen von Lungengewebebiopsieproben von IPAH-Patienten zeigten eine perivaskuläre Entzündungszellinfiltration von T-Zellen, B-Zellen und Makrophagen
52
,
53
. Austinet al. stellten ferner fest, dass die Zahl der CD8-T-Zellen im Lungengewebe von IPAH-Patienten signifikant erhöht war und die durch abnormale Immunfunktion und Verlust der Autoimmunität verursachte Entzündung mit der Pathophysiologie von IPAH in Zusammenhang stand
54
. Gemäß unseren Ergebnissen haben IPAH-Patienten höhere Konzentrationen von Gedächtnis-B-Zellen, CD8-T-Zellen, follikulären Helfer-T-Zellen, Monozyten und M1- und M2-Makrophagen und niedrigere Konzentrationen von Plasmazellen, gedächtnisruhenden CD4-T-Zellen, Tregs, ruhende NK-Zellen, NK-Zellen, ruhende Mastzellen und Eosinophile. Unsere Ergebnisse stimmen mit denen früherer Studien überein. Daher könnte die Erforschung der Immun- und Stoffwechselmechanismen von IPAH eindeutig den Weg für die Diagnose und Behandlung von IPAH ebnen. Vor allem die Berücksichtigung von Stoffwechselstörungen und Autoimmunität ist entscheidend für die Erforschung der Pathophysiologie von IPAH und den Abbau von Therapeutika. Das metabolische Syndrom ist ein klinisches Merkmal, das hauptsächlich durch Stoffwechselstörungen gekennzeichnet ist. Die beiden Krankheiten sind eng miteinander verbunden,In den letzten Jahren ist es für Mediziner zu einem Trend geworden, Bioinformatik-Technologie, maschinelle Lernalgorithmen und Deep-Learning-Methoden einzusetzen, um verwandte medizinische Probleme zu lösen, und es gibt unzählige verwandte Literatur. Wissenschaftler haben einige fortschrittliche Computermodelle zur Analyse bestehender lncRNA-Krankheits-Assoziationen und zur Vorhersage potenzieller menschlicher lnc-RNAs für Krankheits-Krankheits-Assoziationen erstellt, die effektiv verwendet werden können, um krankheitsassoziierte lnc-RNAs in großem Umfang zu identifizieren und die vielversprechendsten krankheitsassoziierten auszuwählen lnc-RNAs für die experimentelle Validierung
55
. Es gibt auch Modelle auf der Grundlage von Netzwerkalgorithmen und Modelle auf der Grundlage von maschinellem Lernen, um neue Circular-RNAs-Berechnungsmodelle für die Korrelation von Krankheiten vorherzusagen
56
. Während traditionelle biologische Experimente normalerweise viel Zeit und Geld erfordern, um die Unterschiede in der Konzentration bestimmter Metaboliten bei Patienten und denen bei gesunden Menschen zu untersuchen, kann ein neuer Deep-Learning-Algorithmus namens Graph Convolutional Network with Graph Attention Network (GCNAT) das Potenzial vorhersagen Assoziationen von krankheitsassoziierten Metaboliten
57
. Advanced Model Design ist in den letzten Jahren häufiger geworden, insbesondere in Form einer sinnvollen Kombination mehrerer Algorithmen, ein Prozess, der als Modellfusion bekannt ist. Die Kombination mehrerer Algorithmen zur Verbesserung der Modellleistung und der Vorhersagekraft ist zum heißesten Trend geworden
58
. Unsere Forschung kombiniert zwei maschinelle Lernalgorithmen, um die Vorhersagefähigkeit von IPAH- und komorbiden MS-Diagnosegenen mit hoher Zuverlässigkeit erheblich zu verbessern.EinschränkungenUnsere Studie hat mehrere Einschränkungen. Obwohl wir zwei IPAH-Datensätze gepoolt haben, blieb die Gesamtzahl der in diese Studie aufgenommenen Proben begrenzt. Obwohl die identifizierten diagnostischen Kandidatengene hauptsächlich bei der Regulierung von Immun- und Stoffwechselwegen angereichert wurden, sind die Wechselwirkungen zwischen diesen diagnostischen Kandidatengenen und dysregulierten Immunzellen noch weitere Untersuchungen wert.FazitUnseres Wissens ist dies die erste Studie, die diagnostische Gene dokumentiert, die gemeinsam mit IPAH und MS assoziiert sind. Wir identifizierten insgesamt 11 immun- und stoffwechselbezogene diagnostische Gene ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1 ) durch verschiedene bioinformatische Analysen und maschinelle Lernalgorithmen, die dann bereitgestellt wurden ein Nomogramm für die Diagnose von MS in Kombination mit IPAH. Wir haben auch darauf hingewiesen, dass ein Teil der IPAH-Immunzellen fehlreguliert ist. Schließlich wurden Unterschiede in der diagnostischen Genexpression anhand von Lungengewebedaten von IPAH-Patienten in der GSE48149-Datenbank validiert.
Der IPAH-Datensatz GSE48149 für Lungengewebe verifiziert die Expression diagnostischer Kandidatengene.
DiskussionDie Ätiologie der IPAH ist unbekannt, dennoch stellt die Krankheit eine große physische, mentale und wirtschaftliche Belastung für die Patienten dar. Bestehende Studien haben einen Teil neuer Biomarker identifiziert, die die Diagnose von IPAH erleichtern können. Über Immuninfiltrationsstudien von IPAH wurde ursprünglich bereits berichtet, aber diese Untersuchung ist die erste, die IPAH mit MS kombiniert. In der Zwischenzeit wurde die Identifizierung von diagnostischen Kandidatengenen bei der Diagnose von IPAH nicht berücksichtigt. Wir haben eine Reihe integrierter bioinformatischer Analysen und maschineller Lernmethoden verwendet, um gemeinsame Signalwege und gemeinsame diagnostische Kandidatengene für IPAH und MS zu identifizieren. Um Fehler zu vermeiden, haben wir die DEGs- und WGCNA-Modulgene kombiniert, um insgesamt 287 gemeinsame Kandidatengene zu identifizieren. Anreicherungsanalysen zeigten, dass diese Kandidatengene mit immun- und stoffwechselbezogenen Signalwegen assoziiert sind. Als nächstes wendeten wir einen maschinellen Lernansatz an, um weiter nach Schlüsselgenen zu suchen. Die Crossover-Ergebnisse von Random-Forest- und LASSO-Analysen wurden als gemeinsame diagnostische Kandidatengene für IPAH und MS betrachtet, und wir haben die diagnostische Wirkung jedes gemeinsamen diagnostischen Kandidatengens weiter validiert. Insbesondere,EVI5L , RNASE2 und PARP10 haben einen großen diagnostischen Wert und hohe AUC-Werte.IPAH ist eine seltene Erkrankung, die durch einen erhöhten Lungengefäßwiderstand gekennzeichnet ist. In dieser Studie haben wir zwei Datensätze mit IPAH-Lungengewebe als Analyseproben ausgewählt, die für die Genexpression bei IPAH-Patienten repräsentativer waren als die Gensequenzierung von peripherem Blut. Wir haben dann die obigen Ergebnisse anhand der Daten verifiziert und festgestellt, dass die identifizierten diagnostischen Kandidatengene in einem anderen IPAH-Lungengewebedatensatz gleichermaßen unterschiedlich exprimiert wurden. Daher könnten wir schlussfolgern, dass die entdeckten diagnostischen Kandidatengene versteckte IPAH durch periphere Blutuntersuchungen von MS-Patienten erkennen können, was eine wahnsinnig einfache und wirtschaftliche Operation ist und eine invasive Untersuchung durch Rechtsherzkatheter vermeidet.Letztendlich identifizierten wir 11 wichtige Kandidatengene ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1 ) und es wurde auch ein Nomogramm zur Diagnose von IPAH bei MS-Patienten erstellt, das einen hohen diagnostischen Wert aufweist.EVI5L gehört zu einer kleinen Unterfamilie von TRE-2/BUB2/CDC16-Domänenproteinen und ist ein Nebenprodukt von EVI5 . EVI5L hat etwa eine zu 70 % ähnliche Identität wie Evi5 . Aufgrund der wenigen vorliegenden Berichte über EVI5L haben wir jedoch hauptsächlich EVI5 analysiert. EVI5 hat verschiedene regulatorische Rollen bei der Zellzyklusprogression, der Zytokinese und dem Transport von Zellmembranen. In Tumoren ist die EVI5- Expression bei mehreren Krebsarten wie nicht-kleinzelligem Lungenkrebs, Kehlkopfkrebs und hepatozellulärem Karzinom fehlreguliert, und EVI5 gilt daher als potenzielle Onkogene und Zellzyklusregulatoren
25
,
26
,
27
.EVI5 ist auch ein Risikofaktor für Multiple Sklerose
28
. Multiple Sklerose ist eine ziemlich häufige demyelinisierende Autoimmunerkrankung; EVI5L könnte daher als immunbezogenes Gen eine wichtige Rolle bei der zellulären Immunität spielen. Der Mechanismus von EVI5L bei IPAH und MS erfordert jedoch weitere Untersuchungen.RNASE2 ist ein von Eosinophilen abgeleitetes Neurotoxin (EDN/RNase2) und eine endolysosomale Ribonuklease, die synergistisch wirkt, um Uridin aus Oligonukleotiden freizusetzen. RNASE2 aktiviert den humanen Toll-like-Rezeptor 8 (TLR8), während die TLR8-Aktivierung eine starke T-Helfer-1-Zellantwort induziert, die für die Abwehr intrazellulärer Pathogene entscheidend ist. Dies deutet darauf hin, dass RNASE2 eine wichtige Rolle im Immunsystem spielt
29
. Als immunbezogenes Molekül ist RNASE2 ein Biomarker für verschiedene Erkrankungen des Immunsystems, darunter chronische myeloische Leukämie, systemischer Lupus erythematodes, rheumatoide Arthritis und multiples Myelom
30
,
31
,
32
,
33
. In Bezug auf Krebs fördert RNASE2 die maligne Progression von Gliomen über den PI3K/Akt-Signalweg
34
. Es ist auch ein immunbezogener Biomarker, der zur Bewertung der Prognose von Magen- und Nierenkrebs verwendet wird
35
,
36
. Im Atmungssystem beeinflusst RNASE2 die Eosinophilen-spezifischen Proteinspiegel der Asthmafamilie und spielt eine Schlüsselrolle bei allergischen Reaktionen, die Asthma auslösen
37
. Frühere bioinformatische Studien haben gezeigt, dass RNASE2 in IPAH überexprimiert wird und ein Biomarker von IPAH ist
38
. Die bestehende Forschung definiert jedoch die Hauptrolle von RNASE2 noch nicht klarim IPAH. In dieser Studie fanden wir heraus, dass RNASE2 ein gemeinsamer immun- und stoffwechselbezogener Biomarker für MS und IPAH ist, was darauf hindeutet, dass RNASE2 für die Entwicklung von Stoffwechselstörungen bei beiden Krankheiten verantwortlich sein könnte, was beweist, dass es eine wichtige potenzielle Rolle bei der Diagnose spielt MS-Patienten mit IPAH.PARP10 , alternativ bekannt als ARTD10 , ist ein Mitglied der PARP-Proteinfamilie, das eine Mono-ADP-Ribosylierung von Zielproteinen durchführt
39
. PARP10 ist ein Stoffwechselregulator, der eine wichtige Rolle im Fettstoffwechsel spielt. Das Silencing von PARP10 induziert mitochondriale Oxidation und AMPK-Aktivität. PARP10 ist an der Regulierung der zellulären Autophagie in Zellmodellen beteiligt; in einem Zellkrebsmodell induziert der Verlust von PARP10 die Fettsäureoxidation
40
. PARP10 wird häufig in menschlichen Geweben exprimiert, insbesondere in Leber und Milz. Die Sekretion von Apolipoprotein B in der Leber ist abhängig von PARP10 , undDie Stummschaltung von PARP10 reduziert die Expression von Apolipoprotein B in menschlichen Hepatozyten
41
. Daher kann die Expression von PARP10 Lipoproteinspiegel sehr niedriger, mittlerer Dichte und niedriger Dichte beeinflussen, und PARP10 ist eng mit dem Lipidstoffwechsel verbunden. PARP10 ist auch an der Entzündungsreaktion und der Tumorentwicklung beteiligt, da es in den meisten menschlichen Tumoren überexprimiert wird, einschließlich Brust- und Eierstocktumoren, oralem Plattenepithelkarzinom, kolorektalem Karzinom und hepatozellulärem Karzinom, und PARP10 spielt auch eine Rolle bei der Förderung der Proliferation von verwandte Tumoren
42
,
43
,
44
,
45
. Darüber hinaus ist PARP10 für Anti-DNA-Schäden erforderlich, und der Knockout des PARP10 -Gens verursacht eine zelluläre Überempfindlichkeit gegenüber DNA-Schäden und einen DNA-Replikationsdefekt
46
. Wir stellten fest, dass die Crossover-Gene von IPAH und MS hauptsächlich in Stoffwechsel- und Immunwegen angereichert sind und stellten fest, dass PARP10 als Stoffwechselregulator eine wichtige Rolle bei der Entstehung und Entwicklung beider Krankheiten spielt. Unsere Studie hat gezeigt, dass die Überexpression von PARP10 bei Patienten mit IPAH mit MS ein lebenswichtiger stoffwechselbezogener Biomarker bei Patienten sein kann und einen hohen diagnostischen Wert hat.Stoffwechselstörungen sind eine wichtige Pathogenese von PAH, und die medikamentöse Behandlung des pathologischen Stoffwechselzustands eines Patienten zur Behandlung von erhöhtem Lungenarteriendruck ist ein Bereich, der von vielen Forschern aktiv untersucht wird. Tiermodelltests ergaben, dass das hypoglykämische Medikament Metformin die Endothelfunktion bei PAH verbesserte und das Überleben von PAH-Ratten signifikant verlängerte
47
. Die Ergebnisse einer klinischen Studie bestätigten auch, dass Biguanid, ein oral verabreichtes hypoglykämisches Medikament, die rechtsventrikuläre Fraktionsfläche von PAH-Patienten signifikant verbesserte, mit einer guten therapeutischen Wirkung
48
. Legtschenko et al. fanden heraus, dass der PPAR-γ-Agonist Pioglitazon die pulmonale Hypertonie durch Fettsäureoxidation umkehrte, die hauptsächlich mit dem Fettstoffwechsel und einer gestörten mitochondrialen Morphologie/Funktion bei rechtsventrikulärem Versagen und pulmonalvaskulärer Hypertonie verbunden war
49
. Der Natrium-Glucose-Cotransporter 2 (SGLT2)-Inhibitor Englizin verbesserte die Glukoseausscheidung im Urin und reduzierte kardiovaskuläre Ereignisse und Mortalität bei Patienten mit Typ-2-Diabetes. In ihrer Studie
50
stellten sie fest, dass SGLT2 die Sterblichkeit bei MCT-induzierten PAH-Ratten reduzierte und den maladaptiven Lungenumbau reduzierte.Entzündung ist eine kritische Komponente aller Subtypen von PAH, aktivierte Immunzellen sezernierten mehr Zytokine, wie z. B. Tumornekrosefaktor-α und Interleukine, und können in den Seren von Patienten in Konzentrationen gefunden werden, die positiv mit der Schwere der Erkrankung bei PAH korrelieren
51
. Es wurde gezeigt, dass viele zirkulierende Immunzellen (z. B. Makrophagen, Monozyten, Mastzellen, dendritische Zellen und T-Zellen) bei PAH in Milz und Lunge aktiviert werden, und eine große Anzahl wird in den Lungenkreislauf rekrutiert oder innerhalb des Lungenkreislaufs aktiviert. Sie regulieren die Funktion und den Differenzierungsstatus der Pulmonalarterienzellen auf parakrine Weise. Die Arten von Immunzellen, die an PAH beteiligt sind, können bei Aktivierung stark glykolytisch werden, was darauf hindeutet, dass diese Zellen möglicherweise auch auf veränderte Stoffwechseltherapien und andere Faktoren ansprechen
51
. Frühere Untersuchungen von Lungengewebebiopsieproben von IPAH-Patienten zeigten eine perivaskuläre Entzündungszellinfiltration von T-Zellen, B-Zellen und Makrophagen
52
,
53
. Austinet al. stellten ferner fest, dass die Zahl der CD8-T-Zellen im Lungengewebe von IPAH-Patienten signifikant erhöht war und die durch abnormale Immunfunktion und Verlust der Autoimmunität verursachte Entzündung mit der Pathophysiologie von IPAH in Zusammenhang stand
54
. Gemäß unseren Ergebnissen haben IPAH-Patienten höhere Konzentrationen von Gedächtnis-B-Zellen, CD8-T-Zellen, follikulären Helfer-T-Zellen, Monozyten und M1- und M2-Makrophagen und niedrigere Konzentrationen von Plasmazellen, gedächtnisruhenden CD4-T-Zellen, Tregs, ruhende NK-Zellen, NK-Zellen, ruhende Mastzellen und Eosinophile. Unsere Ergebnisse stimmen mit denen früherer Studien überein. Daher könnte die Erforschung der Immun- und Stoffwechselmechanismen von IPAH eindeutig den Weg für die Diagnose und Behandlung von IPAH ebnen. Vor allem die Berücksichtigung von Stoffwechselstörungen und Autoimmunität ist entscheidend für die Erforschung der Pathophysiologie von IPAH und den Abbau von Therapeutika. Das metabolische Syndrom ist ein klinisches Merkmal, das hauptsächlich durch Stoffwechselstörungen gekennzeichnet ist. Die beiden Krankheiten sind eng miteinander verbunden,In den letzten Jahren ist es für Mediziner zu einem Trend geworden, Bioinformatik-Technologie, maschinelle Lernalgorithmen und Deep-Learning-Methoden einzusetzen, um verwandte medizinische Probleme zu lösen, und es gibt unzählige verwandte Literatur. Wissenschaftler haben einige fortschrittliche Computermodelle zur Analyse bestehender lncRNA-Krankheits-Assoziationen und zur Vorhersage potenzieller menschlicher lnc-RNAs für Krankheits-Krankheits-Assoziationen erstellt, die effektiv verwendet werden können, um krankheitsassoziierte lnc-RNAs in großem Umfang zu identifizieren und die vielversprechendsten krankheitsassoziierten auszuwählen lnc-RNAs für die experimentelle Validierung
55
. Es gibt auch Modelle auf der Grundlage von Netzwerkalgorithmen und Modelle auf der Grundlage von maschinellem Lernen, um neue Circular-RNAs-Berechnungsmodelle für die Korrelation von Krankheiten vorherzusagen
56
. Während traditionelle biologische Experimente normalerweise viel Zeit und Geld erfordern, um die Unterschiede in der Konzentration bestimmter Metaboliten bei Patienten und denen bei gesunden Menschen zu untersuchen, kann ein neuer Deep-Learning-Algorithmus namens Graph Convolutional Network with Graph Attention Network (GCNAT) das Potenzial vorhersagen Assoziationen von krankheitsassoziierten Metaboliten
57
. Advanced Model Design ist in den letzten Jahren häufiger geworden, insbesondere in Form einer sinnvollen Kombination mehrerer Algorithmen, ein Prozess, der als Modellfusion bekannt ist. Die Kombination mehrerer Algorithmen zur Verbesserung der Modellleistung und der Vorhersagekraft ist zum heißesten Trend geworden
58
. Unsere Forschung kombiniert zwei maschinelle Lernalgorithmen, um die Vorhersagefähigkeit von IPAH- und komorbiden MS-Diagnosegenen mit hoher Zuverlässigkeit erheblich zu verbessern.EinschränkungenUnsere Studie hat mehrere Einschränkungen. Obwohl wir zwei IPAH-Datensätze gepoolt haben, blieb die Gesamtzahl der in diese Studie aufgenommenen Proben begrenzt. Obwohl die identifizierten diagnostischen Kandidatengene hauptsächlich bei der Regulierung von Immun- und Stoffwechselwegen angereichert wurden, sind die Wechselwirkungen zwischen diesen diagnostischen Kandidatengenen und dysregulierten Immunzellen noch weitere Untersuchungen wert.FazitUnseres Wissens ist dies die erste Studie, die diagnostische Gene dokumentiert, die gemeinsam mit IPAH und MS assoziiert sind. Wir identifizierten insgesamt 11 immun- und stoffwechselbezogene diagnostische Gene ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1 ) durch verschiedene bioinformatische Analysen und maschinelle Lernalgorithmen, die dann bereitgestellt wurden ein Nomogramm für die Diagnose von MS in Kombination mit IPAH. Wir haben auch darauf hingewiesen, dass ein Teil der IPAH-Immunzellen fehlreguliert ist. Schließlich wurden Unterschiede in der diagnostischen Genexpression anhand von Lungengewebedaten von IPAH-Patienten in der GSE48149-Datenbank validiert.
www.nature.com/articles/s41598-023-27435-4
AbstraktIdiopathische pulmonale Hypertonie (IPAH) ist eine Erkrankung, die verschiedene Gewebe und Organe sowie das Stoffwechsel- und Entzündungssystem betrifft. Die am weitesten verbreitete Stoffwechselerkrankung ist das metabolische Syndrom (MS), das Insulinresistenz, Dyslipidämie und Fettleibigkeit umfasst. Basierend auf einer Fülle von Studien kann es einen Zusammenhang zwischen IPAH und MS geben, obwohl die zugrunde liegende Pathogenese unklar bleibt. Durch verschiedene bioinformatische Analysen und maschinelle Lernalgorithmen identifizierten wir 11 immun- und stoffwechselbezogene potenzielle diagnostische Gene ( EVI5L, RNASE2, PARP10, TMEM131, TNFRSF1B, BSDC1, ACOT2, SAC3D1, SLA2, P4HB und PHF1) für die Diagnose von IPAH und MS, und wir liefern hier ein Nomogramm für die Diagnose von IPAH bei MS-Patienten. Außerdem haben wir die abweichenden Immunzellen von IPAH entdeckt und diskutieren sie hier.EinführungDie pulmonale arterielle Hypertonie (PAH) ist eine seltene Erkrankung, die durch den Verschluss von Arteriolen in der Lunge gekennzeichnet ist, was zu einem deutlichen Anstieg des pulmonalen Gefäßwiderstands führt 1 . Es gibt viele Risikofaktoren für das Auftreten von PAH, darunter Stoffwechselstörungen, Hyperlipidämie, Fettleibigkeit, Insulinresistenz, fehlregulierte Proliferation von Gefäßzellen, abnormaler Zellstoffwechsel, Entzündungen und Genmutationen 2 . Idiopathische PAH (IPAH) ist eine wichtige Art von PAH, deren klinische Symptome nicht spezifisch sind; Die Patienten zeigen hauptsächlich Symptome im Zusammenhang mit einer fortschreitenden Funktionsstörung des rechten Herzens, die oft durch Müdigkeit verursacht wird, und zeigen Müdigkeit, Dyspnoe, Engegefühl in der Brust, Schmerzen in der Brust und Synkopen. IPAH wird durch mehrere pathogene Faktoren produziert, aber der spezifische pathogene Mechanismus wurde nicht vollständig aufgeklärt. Das metabolische Syndrom (MS) wird durch eine Reihe von kardiovaskulären und metabolischen Risikofaktoren ausgelöst, die miteinander in Verbindung stehen. Zu den Risikofaktoren gehören Stoffwechselstörungen, Bluthochdruck, Insulinresistenz, Glukoseintoleranz, zentrale Fettleibigkeit, Dyslipidämie und entzündliche Effekte 3 . Die Pathogenese der beiden Krankheiten weist Gemeinsamkeiten auf, und Patienten mit MS haben ein höheres Risiko für eine Anfälligkeit für IPAH.IPAH gilt als systemische Erkrankung und betrifft viele Organe und Gewebe sowie die Entzündungs- und Stoffwechselwege 4 . Die Rolle des knochenmorphogenen Proteinrezeptors Typ 2 (BMPR2) und vieler seiner nachgeschalteten Ziele, wie z. B. Peroxisom-Proliferator-aktivierter Rezeptor (PPAR)-γ und Apolipoprotein E, bei der Induktion der IPAH-Produktion über den Stoffwechselweg wurden ausführlich beschrieben, und PPAR -γ und Apolipoprotein E stehen ebenfalls in Zusammenhang mit einer Vielzahl von pathologischen Stoffwechselzuständen 5 . Stoffwechselstörungen sind seit langem bei IPAH weit verbreitet, und eine zunehmende Zahl von Studien deutet auf eine starke Verbindung zwischen IPAH und MS hin; Die beiden Krankheiten wurden jedoch nie zusammen untersucht. Daher wird diese Studie den Zusammenhang zwischen den beiden Krankheiten untersuchen und gemeinsame Biomarker für die Diagnose von Krankheiten aufdecken.Die bioinformatische Analyse hilft uns, die Ätiologie der Krankheit zu erforschen, während die Gen-Microarray-Technologie neue Ideen zur Erforschung der Pathogenese von IPAH und MS liefert. In dieser Studie haben wir maschinelle Lernalgorithmen für bioinformatische Analysen kombiniert, um diagnostische Kandidatengene und Signalwege zu identifizieren, die von IPAH und MS aus Gene Expression Omnibus-Datensätzen geteilt werden. Dies ist auch die erste Studie, die auf die gemeinsamen Biomarker und verwandte Stoffwechselwege von IPAH und MS abzielt, eine diagnostische Genexpressionsvalidierung wurde in einem anderen GEO-Datensatz durchgeführt. Unsere Studie liefert neue Erkenntnisse für die Erforschung der genetischen Ätiologie und Kombinationsbehandlungsstrategien für IPAH- und METS-Komorbidität. Darüber hinaus untersuchten wir auch die Infiltration von Immunzellen bei IPAH. Materialen und Methoden.DatenverarbeitungAus der Gene Expression Omnibus - Datenbank ( https : //www.ncbi.nlm .nih.gov/geo/ ) 9 . Die GSE15197-Serie umfasste Proben von 13 Kontrollgruppen und 18 IPAH-Patientengruppen und die GSE117261-Serie umfasste Proben von 20 Kontrollgruppen und 32 IPAH-Patientengruppen, während die GSE98895-Serie Proben von 20 Kontrollgruppen und 20 MS-Patientengruppen umfasste. Die IPAH-Datenbankproben wurden aus menschlichem Lungengewebe und die MS-Datenbankproben aus peripherem Blut von Patienten gewonnen. Ein einzelnes Validierungsset, GSE48149 10 , das 17 Lungengewebeproben von 8 IPAH-Patientengruppen und 9 normalen Kontrollen enthielt, wurde ebenfalls verwendet. Um Studienfehler zu vermeiden, haben wir Geschlecht und Alter zwischen Patienten und gesunden Kontrollen in den Datensätzen ausgeschlossen. Einzelheiten zu diesen Datensätzen sind in der Ergänzungstabelle S1 enthalten . Das Flussdiagramm dieser Studie ist in Abb. 1 dargestellt , gezeichnet vom WPS-Büro 11 (Kingsoft, China, Version: 11.1.0.11754).Abbildung 1









OMNIA TEMPUS HABENT
Diagnose IPAH im Februar 2013, in Behandlung bei OA Dr. Ulrich Krüger, jetzt Dr. Fischer Herzzentrum Duisburg, Medikamente: Sildenafil, Bosentan jetzt Macitentan, Subkutane Treprostinilpumpe, seit Januar 2024 getunnelter ZVK mit externer Pumpe (Groshongkatheter), 24/7 Sauerstoff, Marcumar, Diuretika
Bitte Anmelden oder Registrieren um der Konversation beizutreten.
